DRUGONE
在药物设计中,源自自然语言处理的 Transformer 网络被广泛用于多种生成与预测任务。研究人员采用基于序列的生成性化合物设计作为模型体系,系统分析了 Transformer 的学习特征,并检验其是否能够学习与蛋白–配体相互作用相关的信息。结果显示,Transformer 模型在预测活性化合物时,需要至少约 60% 的原始测试序列才能维持性能。预测结果依赖于训练与测试数据之间的序列相似性、化合物相似性以及模型对训练化合物的记忆效应。这些预测完全由统计关联驱动,通过将序列模式与分子结构进行映射来实现,而非学习到生物学相关的信息。研究结果提示,虽然基于序列的化合物设计具有应用潜力,但在解释 Transformer 模型的结果时应保持谨慎。

Transformer 模型源自自然语言处理领域,如今已被广泛应用于化学与药物发现等科学研究中,被称为化学语言模型(Chemical Language Models, CLMs)。这些模型以分子结构的文本表示(如 SMILES 字符串)为输入,通过注意力机制在生成与预测任务中取得优异性能。Transformer 的自注意力机制使其在许多任务中超越了传统神经网络结构,被用于化合物生成、性质预测等多种应用。然而,这类模型的预测难以解释,限制了其在交叉领域研究中的可解释性与可接受度。
在药物发现中,Transformer 的灵活结构与强大的学习能力使其能够应对非传统的生成性设计任务,特别是从蛋白质序列直接生成活性化合物的尝试。早期的机器学习研究曾探索如何利用蛋白序列和配体信息预测蛋白–配体相互作用,但直接从序列生成活性化合物的研究极为有限。相比于三维结构驱动的药物设计,基于序列的设计缺乏明确的科学依据,因为蛋白质中参与配体结合的残基数量有限,序列相似性并不总能反映相同的结构或结合特征。
自 Transformer 引入化学领域以来,已有多项研究报告了基于序列的化合物生成,证明了 Transformer 能够从序列到化合物进行映射并生成活性分子。这些研究多采用“预训练–微调”策略,先在大规模序列–化合物配对数据上训练模型,再针对特定靶标或家族进行微调。然而,对于这些模型成功生成活性化合物的内在机制,目前仍缺乏清晰解释。
因此,研究人员在本研究中以基于序列的化合物生成为模型体系,系统分析 Transformer 的学习行为,揭示其预测机制是否真正依赖于蛋白序列中的生物学相关信息。
方法与结果
方法框架
研究人员使用经典 Transformer 架构对大规模的蛋白–化合物序列配对进行预训练,采用不同的数据划分策略(基于序列或蛋白家族)以分析预测依赖性。模型的性能通过其是否能再现已知活性化合物及其核心骨架结构来评估。随后,针对两类主要药物靶标家族——CMGC 蛋白激酶和 GPCR 家族——进行了微调,并分析了多靶化合物(MT compounds)对预测结果的影响。
化合物与核心结构的可复现性
在基于序列划分的预训练模型中,模型能够再现平均每个序列约 2.0 个化合物和 2.8 个核心结构;而基于家族划分的模型几乎无法再现任何测试化合物。进一步分析表明,这种再现性取决于训练与测试数据中是否存在相似序列及其对应的化合物。当测试序列与训练序列相关时,Transformer 能较好地重建目标化合物,否则表现显著下降。
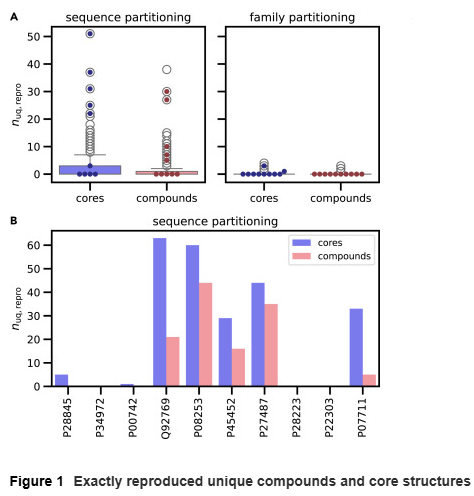
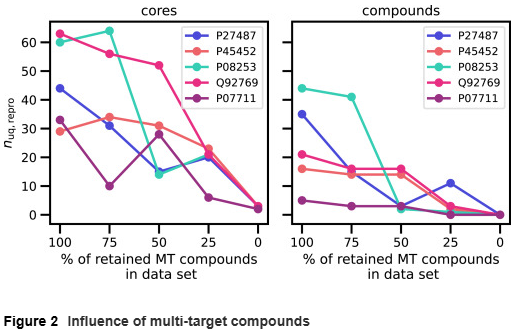
多靶化合物(MT compounds)的影响
约 29% 的数据为多靶化合物,每个平均对应 2.9 条序列。研究人员逐步移除这些多靶化合物后发现,模型再现的测试化合物与核心数量显著下降,当所有多靶化合物被移除时,模型完全无法再现任何测试化合物。这表明 Transformer 在训练过程中记忆了化合物结构,并在测试阶段对相似序列生成相似分子,而非基于序列信息学习到化学或生物学规律。
进一步分析发现,即使模型再现的新化合物在训练集中未出现,其与最近邻训练化合物的结构相似度(Tanimoto 系数)平均仍高达 0.84,说明大多数生成分子源于训练数据的记忆。
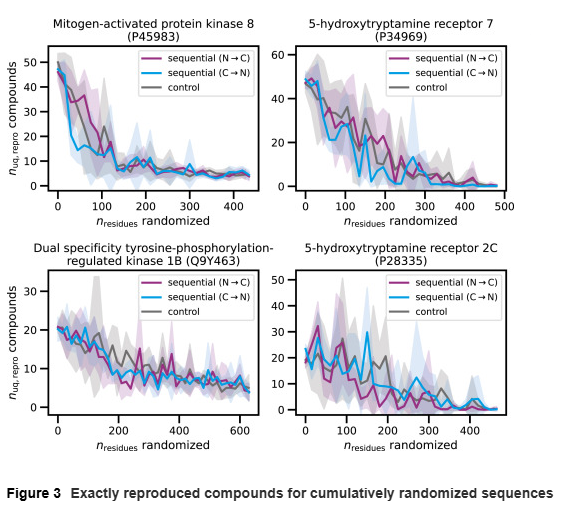
序列随机化实验
研究人员通过逐段随机化测试序列(每次 15 个氨基酸残基)来评估模型对序列扰动的敏感性。结果显示,随着随机化残基比例的增加,模型再现的化合物数量显著减少。当随机化达到约 150–200 个残基时,约一半的测试化合物仍能被再现;但当随机化覆盖整个序列时,模型几乎无法生成原始化合物。
值得注意的是,即便完全随机化 CMGC 激酶序列,模型仍能生成少量化合物,且这些化合物全部属于多靶化合物,进一步印证了记忆效应的主导作用。
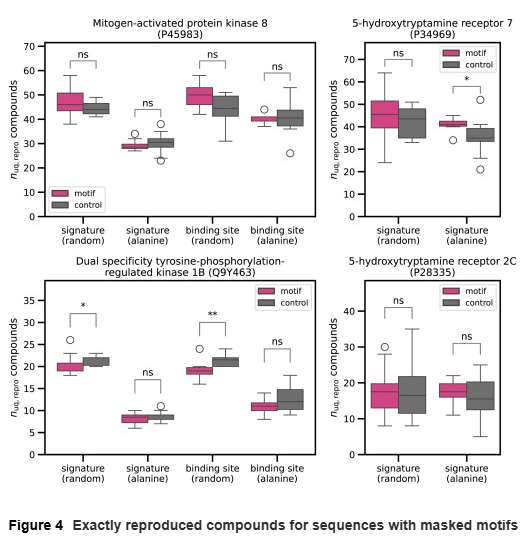
序列基序遮蔽实验
研究人员随机化或用丙氨酸替换关键序列基序(如 GPCR 家族保守区段、激酶的 ATP 结合位点等),以检验模型是否依赖这些生物学相关序列。结果表明,在 12 个测试实例中,有 9 个未显示显著差异,说明模型并未真正学习到特定基序。即便个别实例出现差异,也多被解释为统计波动而非实际的生物学效应。
讨论
研究揭示了 Transformer 在序列到化合物映射中的学习机制:
模型性能高度依赖训练与测试数据的相似性。 当序列或化合物相似性降低时,模型预测能力显著衰减。
化合物记忆主导生成行为。 模型在训练中记住化合物结构,在测试中对相似输入“回忆”出相似分子。
模型未学习生物学特征。 即使遮蔽保守基序或结合位点,预测结果基本不变。
预测为统计驱动,而非机制学习。 模型只是发现序列–化合物的共现模式,而非学习蛋白–配体相互作用。
这种统计式预测虽然在机器学习意义上有效,但若被误解为“模型理解了生物机制”,则可能产生“Clever Hans”式误判——即模型做出正确预测却并未理解背后的因果逻辑。研究人员因此强调,应谨慎解读基于 Transformer 的序列驱动化合物设计结果,并避免将统计相关性误认为机制关联。
结论
研究人员通过系统控制与扰动实验发现:
基于序列的 Transformer 生成模型并未学习蛋白序列中的生物学规律。
其生成行为主要由训练数据的统计相关性和化合物记忆效应驱动。
在解释 Transformer 的生成化合物时,应避免将统计模式误解为生物机制。
该研究为理解生成式化学语言模型的学习机制提供了重要参考,也为未来开发具备真正可解释性和机制感知能力的模型指明了方向。
整理 | DrugOne团队
参考资料
Roth and Bajorath, Unraveling learning characteristics of transformer models for molecular design, Patterns (2025)
https://doi.org/10.1016/j.patter.2025.101392

内容为【DrugOne】公众号原创|转载请注明来源
内容中包含的图片若涉及版权问题,请及时与我们联系删除

