DRUGONE
序列特异性的 DNA 结合蛋白在生物学和生物技术中具有关键作用。研究人员提出了一种计算方法,能够设计出小型 DNA 结合蛋白(DBPs),它们通过在 DNA 大沟中的碱基相互作用识别短的特定序列。该方法成功生成了针对五个不同 DNA 靶点的结合分子,亲和力达到纳摩尔水平。单个结合模块在多达六个碱基对位置上表现出与计算模型高度一致的特异性,并可通过 RFdiffusion 将多个结合模块沿 DNA 双螺旋精确排列,从而实现更高层次的特异性。所设计的蛋白质与 DNA 靶点复合物的晶体结构与模型高度吻合,并且这些蛋白能够在大肠杆菌和哺乳动物细胞中实现邻近基因的转录抑制或激活。这一方法为基因调控和编辑提供了小型、易于递送的序列特异性结合工具。

DNA 结合蛋白在基因调控和基因组编辑中广泛应用,但天然蛋白的结合特异性难以预测,且常受蛋白质骨架构象限制。传统的重新编程方法主要依赖天然蛋白与 DNA 的复合物作为起点,因此局限性明显。相比之下,研究人员希望通过 计算设计,生成能够精准识别任意目标序列的小型模块化蛋白,以补充现有的锌指(ZF)、TALE 和 CRISPR–Cas 等工具。若能实现,将大幅扩展基因调控、基因编辑和诊断中的应用前景。
结果
设计策略
研究人员利用深度学习预测的庞大螺旋-转角-螺旋(HTH)结构库,并结合 RIFdock 蛋白-DNA 对接方法,生成了超过 2.6 万个骨架。随后通过序列设计(Rosetta 和 LigandMPNN)优化界面,使特定氨基酸侧链与 DNA 碱基形成氢键。为提升精度,还引入了侧链预组织化原则,以减少非特异性相互作用。最终,针对每个目标序列生成了上万种候选设计,并筛选出具有最佳能量和氢键网络的分子进入实验验证。
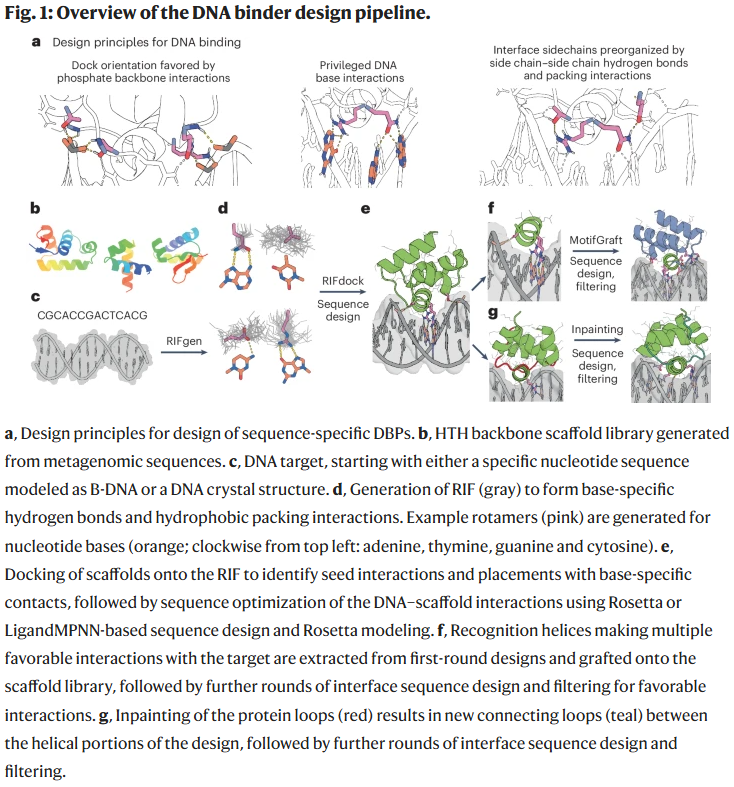
酵母展示与筛选
研究人员通过酵母表面展示体系对设计的蛋白进行筛选,从数万种候选分子中鉴定出 97 个在靶标 DNA 上显著富集的设计。进一步验证发现,44 个候选能够有效结合靶标 DNA,且通过突变关键氨基酸位点可完全破坏结合能力,证明了结合界面的合理性。
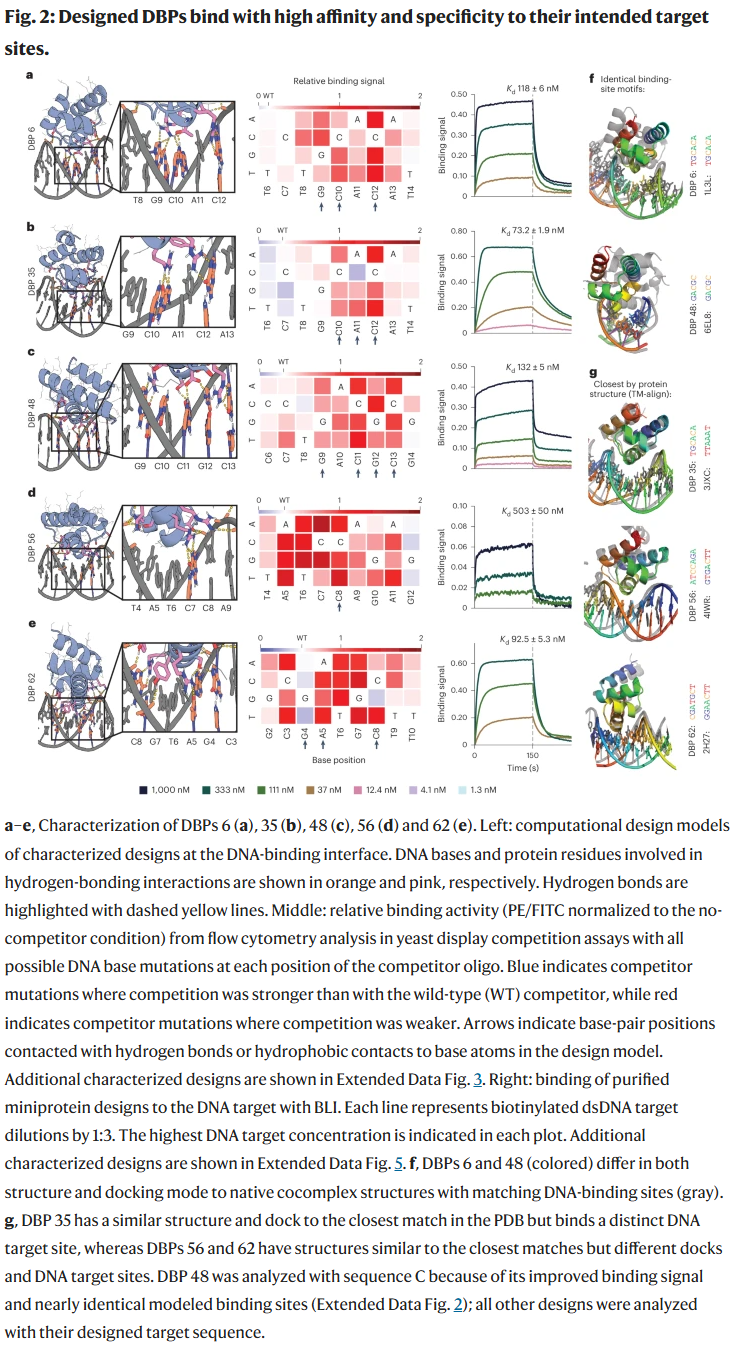
结合特异性与亲和力
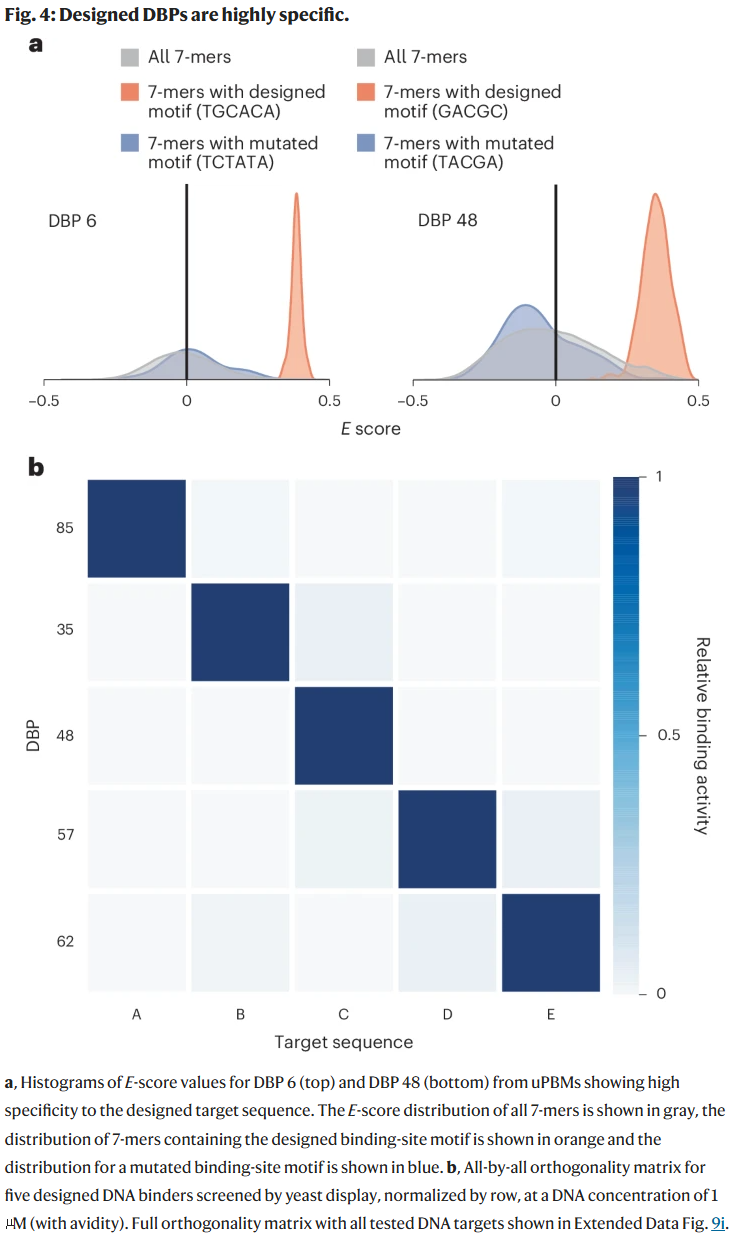
部分设计在结合实验中展现出极高的特异性,与计算模型预测高度一致。例如,DBP6、35、48 等在纳摩尔水平展现出对目标序列的选择性结合,甚至能准确区分单个碱基差异。进一步的生物物理实验表明,这些设计的结合亲和力在 30–500 nM 范围内,接近天然蛋白质水平。
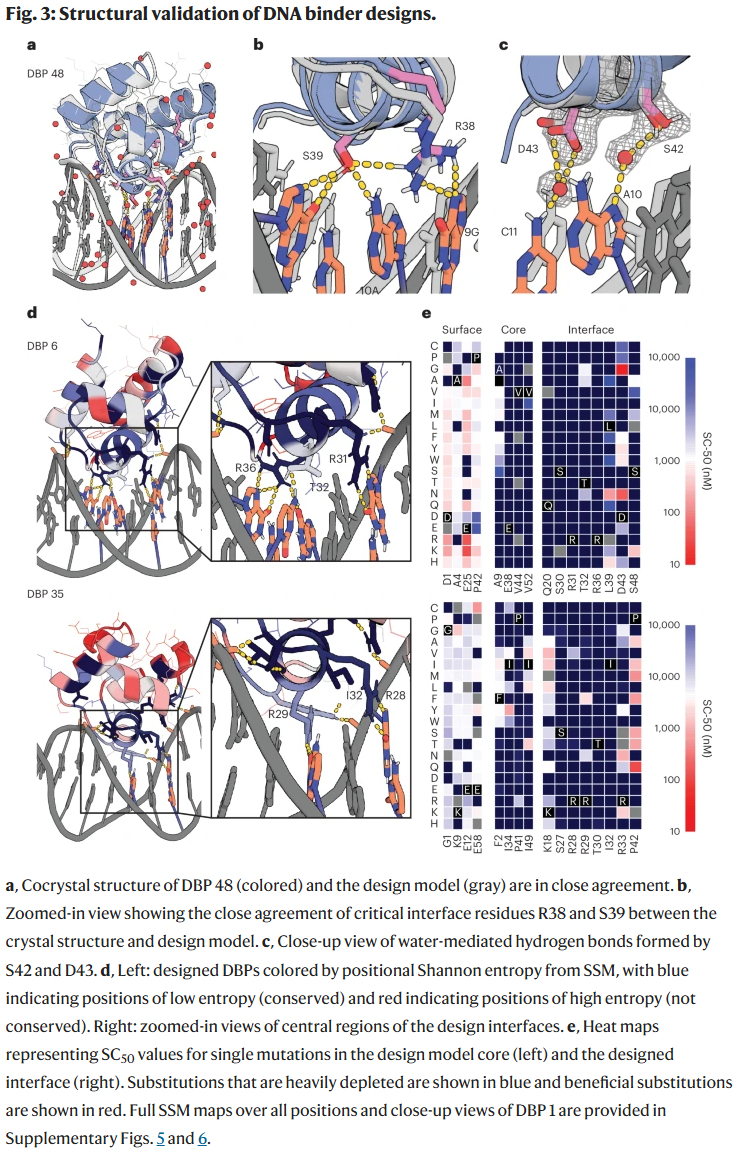
晶体结构验证
研究人员解析了 DBP48 与其靶点序列的复合物晶体结构。结果显示,其整体构象与计算模型高度一致,关键氨基酸-碱基的氢键相互作用得到实验验证,进一步支持了计算设计的准确性。
体内功能验证
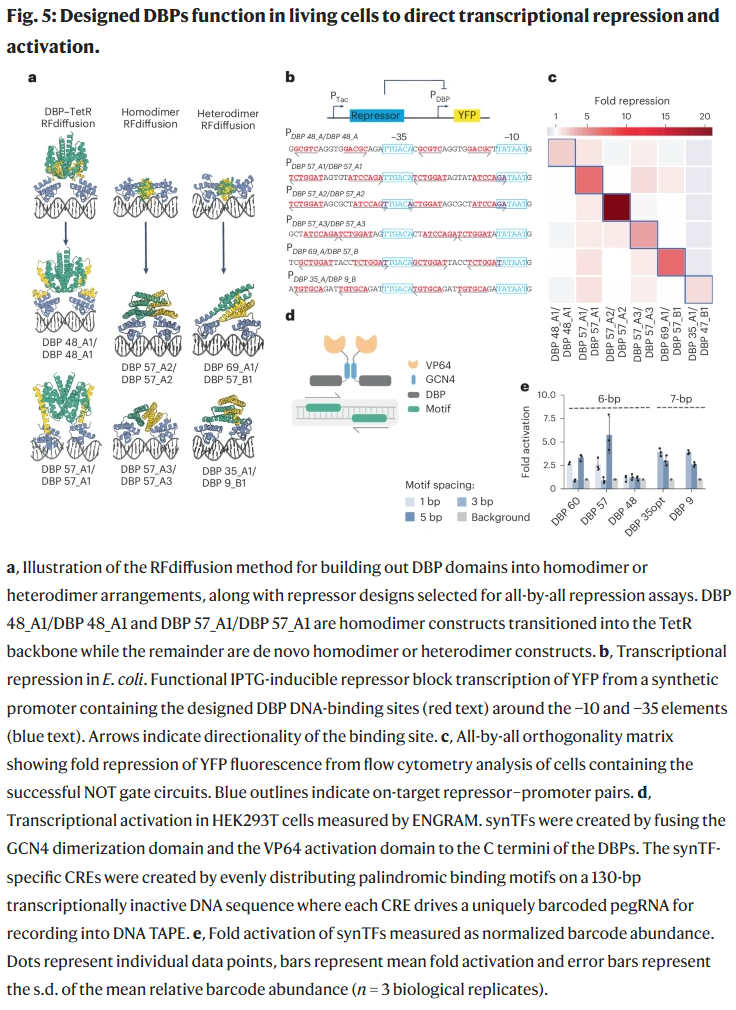
在大肠杆菌中,设计的 DBPs 可作为转录抑制子,阻断报告基因的表达;在哺乳动物细胞中,融合了激活结构域的 DBPs 可实现靶基因启动子的转录激活。这表明这些小型设计蛋白能够在活细胞中有效发挥作用,并具备应用潜力。
讨论
研究人员提出的计算设计方法能够生成小型的序列特异性 DNA 结合蛋白,突破了天然骨架结构的限制。与传统方法相比,这一策略能够:
更高效地生成可递送的小型蛋白;
提高结合位点的特异性与可控性;
在原核与真核细胞中均展现功能性;
可通过模块化方式进一步提升特异性和功能扩展。
未来,该方法可扩展至其他类型的 DNA 结合蛋白骨架,并结合 DNA 构象变化与水介导氢键的建模,从而实现更广泛的应用。研究人员认为,这一策略将在合成生物学、基因治疗和基因编辑等领域发挥重要作用。
整理 | DrugOne团队
参考资料
Glasscock, C.J., Pecoraro, R.J., McHugh, R. et al. Computational design of sequence-specific DNA-binding proteins. Nat Struct Mol Biol (2025).
https://doi.org/10.1038/s41594-025-01669-4

内容为【DrugOne】公众号原创|转载请注明来源
内容中包含的图片若涉及版权问题,请及时与我们联系删除

