DRUGONE
AlphaFold 与大型语言模型的兴起推动了人工智能在分子生物学中的广泛应用。得益于蛋白质数据库和基因组序列数据库中大量的进化信息,研究人员能够利用生成式 AI 进行蛋白质结构预测与设计、构象集合生成以及功能位点识别。这些方法不仅加速了结构预测,也为探索蛋白质的动态行为和进化适应性提供了新思路。本文重点介绍了生成式 AI 在利用进化信息方面的最新进展,涵盖蛋白质设计、构象多样性建模及变构机制预测。

虽然 AI 在蛋白质研究中的应用并非全新领域,但 AlphaFold 的成功极大激发了学界兴趣。研究人员逐渐将大型语言模型与生成式 AI 融入到蛋白质科学中,通过学习进化统计规律来生成新序列或完整原子结构,从而揭示序列—结构—功能之间的深层联系。构象多样性是蛋白质功能与可进化性的核心因素,而生成模型的应用为探索动态过程(如变构、催化和组装)提供了新机遇。然而,如何全面捕捉驱动这些现象的动力学仍是挑战。本文综述了生成式 AI 的最新发展,并特别强调了进化信息在模型构建中的作用。
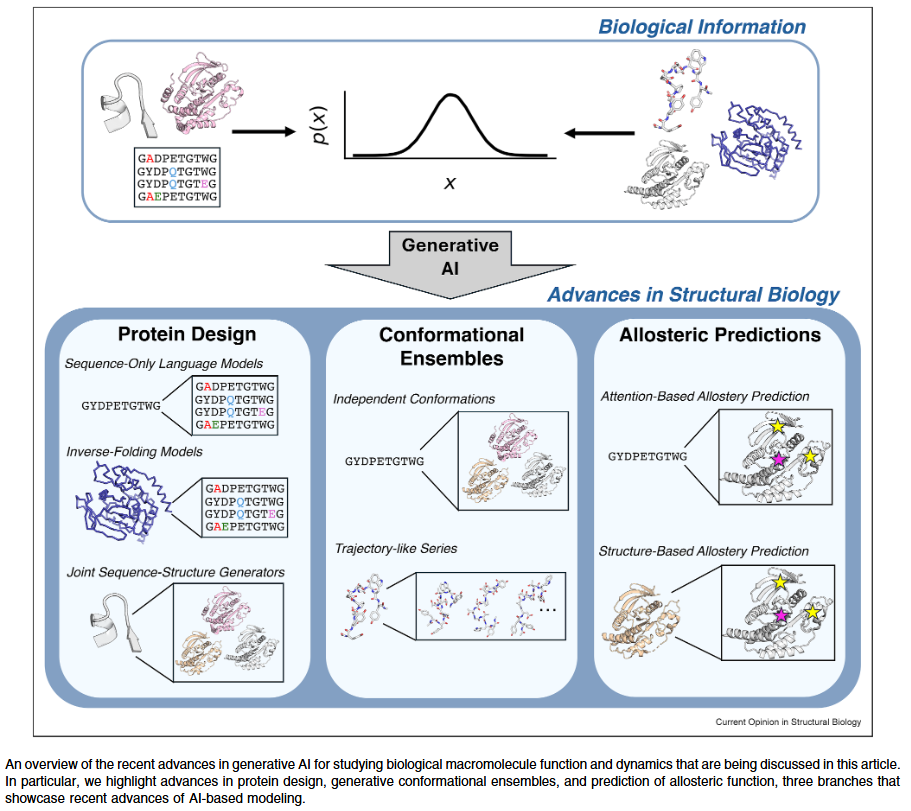
进化:蛋白质设计的信息先验
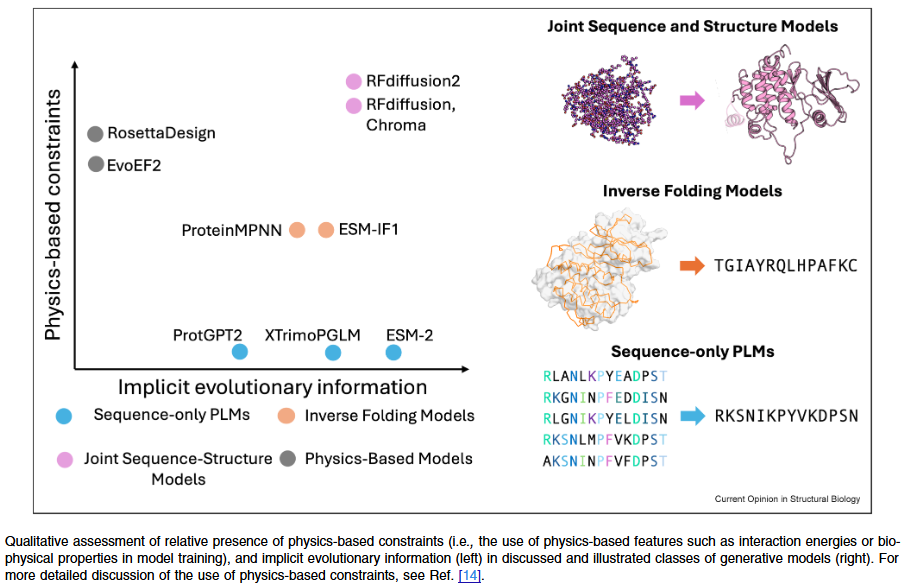
大型蛋白质语言模型通过学习氨基酸之间的统计关系,从序列中捕捉结构和功能模式。它们能够预测合理的突变位点或生成多样化的变体,并在与实验适配时加速定向进化。然而,数据不均衡和模型偏差可能导致过拟合,且训练成本高昂。
MSA Transformer 能显式学习残基共进化关系和系统发育特征,在结构接触预测中优于传统模型。反折叠网络则从结构反推出与之兼容的序列,被广泛用于新型蛋白质设计,如 ProteinMPNN 与 LigandMPNN。这些方法在速度和精度上均展现优势,但仍存在生成多样性不足的问题。
联合生成模型通过扩散或流匹配等方法,同时生成骨架结构和兼容序列,并在训练过程中引入进化正则化。代表性模型如 RFDiffusion、Chroma、OriginFlow,能够探索更广阔的折叠空间,但计算代价极高。
与依赖进化统计的方法不同,物理驱动模型通过能量函数指导序列优化,例如 RosettaDesign 和 EvoEF2。这类方法能够探索非天然氨基酸组合或金属配位体系,但可能生成不物理合理的序列,需要额外的分子动力学或实验验证。
蛋白质构象多样性的预测
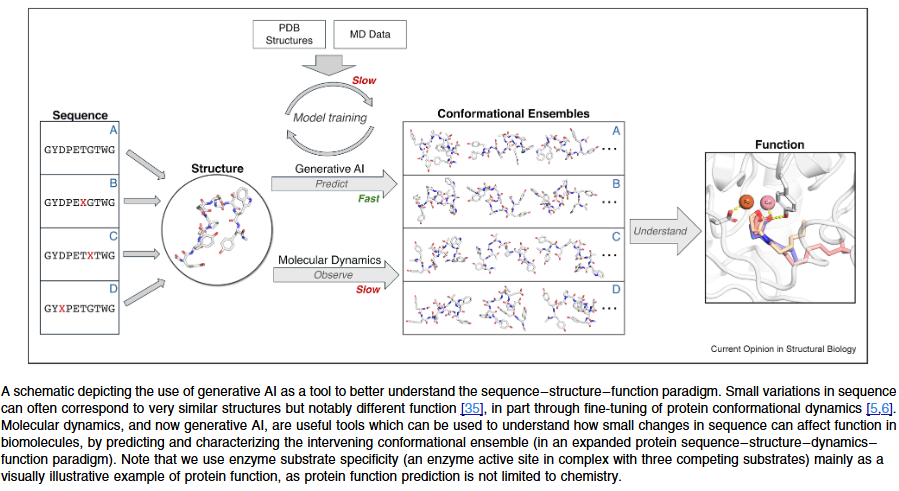
动力学轨迹预测
部分生成式模型尝试从已有状态推断后续构象轨迹,如分子动力学语言模型(MDLM)和扩散模型 EigenFold。这些方法能够以更低计算成本模拟动力学过程,但仍依赖有限训练数据。
构象集合预测
另一类方法聚焦于构象集合生成。BioEmu 结合 MD 与结构数据生成多样化状态;AlphaFlow、ESMFlow 基于流匹配重现 MD 分布;STR2STR 与 AlphaPPImd 则拓展到蛋白质复合物。总体而言,这些模型能以低成本生成接近 MD 水平的构象集合,为功能研究提供了重要补充。
蛋白质变构预测
注意力机制驱动的预测
变构作用在蛋白质功能调控中至关重要。研究人员利用蛋白质语言模型中的注意力分数推断残基间关系,成功预测变构位点。这一方法无需额外训练,且性能优于传统方法。未来工作可进一步结合结构信息和能量函数,提高预测的准确性与可解释性。
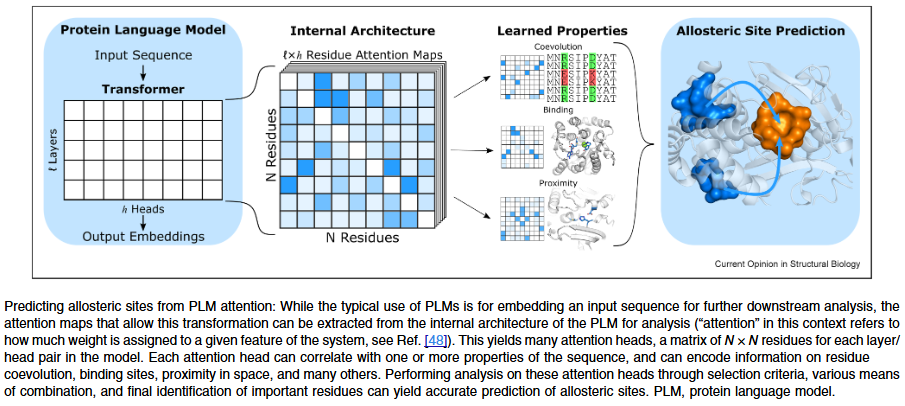
未来方向
PLM 注意力作为无偏指标,展现出识别残基间关系的巨大潜力。随着蛋白质结构与基因组数据的持续积累,未来研究将推动 PLM 在变构预测中的广泛应用,并有望整合至更大规模的功能研究框架。
结论
生成式 AI 在蛋白质科学中的应用快速发展。研究人员已经展示了其在三方面的潜力:
利用进化信息与生成模型推动蛋白质设计;
生成构象集合以解析蛋白质动态;
揭示变构作用与功能调控机制。
虽然模型依赖训练数据,可能存在偏差甚至“幻觉”,但这些局限也能转化为设计上的新机会。未来方向包括从静态结构推断动力学特征、从动力学特征预测功能,以及将网络模型应用于功能映射。总体而言,这一领域发展迅猛,正在为蛋白质研究开辟全新路径。
整理 | DrugOne团队
参考资料
Brownless, A. L. R., Yehorova, D., Welsh, C. L., & Kamerlin, S. C. L. (2025). Generative AI techniques for conformational diversity and evolutionary adaptation of proteins. Current Opinion in Structural Biology, 94, 103135.

内容为【DrugOne】公众号原创|转载请注明来源
内容中包含的图片若涉及版权问题,请及时与我们联系删除

