该论文介绍 DOLPHIN:一种基于深度学习的单细胞转录组分析工具,首次系统性地整合了外显子测序片段(exon reads)和跨剪接点测序片段(junction reads)信息。DOLPHIN 为每个基因构建表征外显子结构的有向图,从而捕捉基因内部的剪接动态(alternative splicing),突破了传统“基因水平”分析的分辨率限制。在多个组织类型和测序平台中,DOLPHIN 显著提升了细胞聚类、剪接事件检测和生物标志物识别的精度,以胰腺癌数据为例,DOLPHIN 识别出大量与患者生存显著相关的外显子级差异信号,而这些信号在传统基因水平分析中被完全掩盖,因而无法被检测到。这种对细胞的高分辨率刻画,能够保留外显子水平的差异和细微的剪接模式,为构建更接近真实分子状态的虚拟细胞提供了坚实基础。高保真度的虚拟细胞不仅可以更准确地模拟疾病在细胞层面的发生和发展,还能帮助研究者揭示关键的致病机制,提升诊断的精准度,并为制定更有针对性的治疗策略提供支持。

单细胞测序是揭示细胞异质性和转录调控机制的重要技术,已经在肿瘤免疫、组织发育和疾病研究等多个领域发挥关键作用。目前主流的单细胞 RNA 测序分析方法通常基于基因水平(gene-level)表达数据,将一个基因的所有转录本的测序片段(reads)汇总为一个总量。然而,这种粗粒度的处理方式掩盖了细胞内大量真实存在的剪接异构性,限制了对细胞状态、功能和疾病相关转录变异的深入理解。在外显子(exon reads)和跨剪接点(junction reads)测序片段中,蕴藏着大量反映剪接调控和转录本结构差异的重要信息。然而,由于数据稀疏、计算挑战大以及缺乏合适的分析框架,这些信息在现有大多数分析流程中被忽略,成为“沉睡的资源”。本研究中,我们提出了 DOLPHIN:一种基于图神经网络的单细胞转录组分析框架,能够系统性整合外显子测序片段和跨剪接点测序片段信息,从而在单细胞层面捕捉更细粒度的剪接结构与表达模式。DOLPHIN 生成的细胞表达表征更加精准、稳定,不仅提升了细胞聚类与亚型识别的分辨率,也为探索疾病相关剪接变异和发现新的分子标志物提供了可能。
方法
基于图结构构建的单细胞表达模型
DOLPHIN 的核心思想是跳出传统的基因总表达量视角,转而从基因内部结构出发,对每个基因在细胞中的外显子和剪接连接关系进行建模。具体来说,DOLPHIN 首先将每个基因构建为一个“剪接有向图”,其中外显子视为图中的节点,剪接位点作为连接这些节点的边;接着将该图嵌入到一个深度学习模型中,学习每个细胞在该基因上的细粒度表达特征。通过整合基因的外显子结构图信息,DOLPHIN 能够生成高分辨率的细胞表征,同时捕捉不同细胞间的剪接异构性和外显子表达差异,这些关键信号在传统的基因水平分析中往往会被掩盖而无法识别。
增强剪接信号的邻域聚合机制
由于单细胞数据中剪接读段稀疏,DOLPHIN 设计了一种基于邻域聚合的机制,用于增强信号。具体来说,模型会自动识别在同一细胞类型中表达相似的“邻居细胞”,并将它们的剪接信息进行汇总,有效提升稀疏区域的信号质量。这一策略确保了即使在低覆盖度(如 10X Genomics)数据中,DOLPHIN 也能稳定提取剪接结构。
统一外显子与剪接位点信息的嵌入表达
DOLPHIN 同时利用外显子表达和剪接连接两个维度的信息,设计了一种统一的嵌入表示方法。在模型中,外显子读段用于捕捉空间位置和表达强度,剪接连接则反映不同转录本之间的结构关系。这种联合建模方式打破了传统“只看表达量”的分析框架,为发现剪接变异、功能外显子和复杂转录事件提供了全新可能。
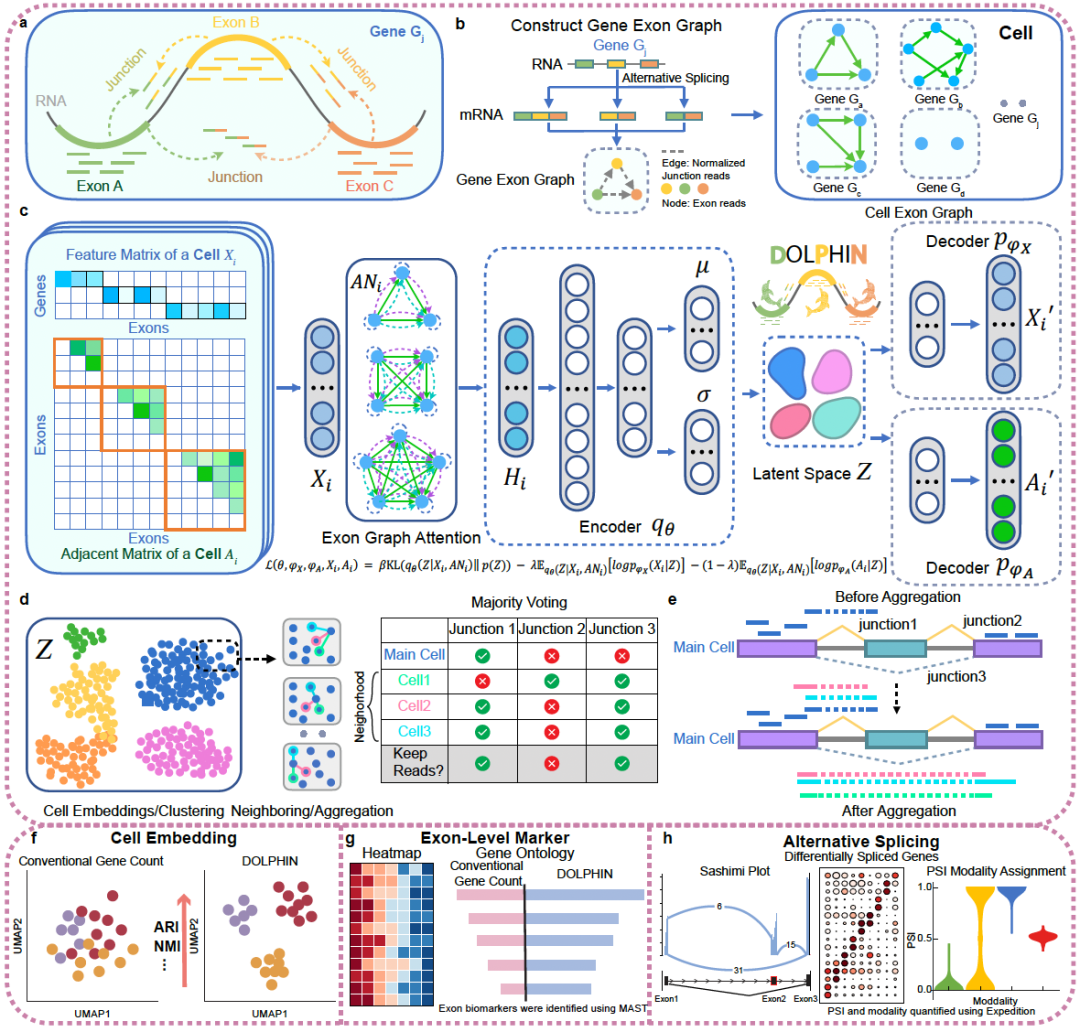
图1 DOLPHIN 方法概览 a, 单细胞 RNA 测序数据的预处理,包括外显子比对读段和外显子-外显子剪接读段的定量。b, 构建基因特异性的外显子图,其中节点表示外显子,边表示剪接连接,最终在每个细胞中形成一张外显子图。c, 通过变分图自动编码器(VGAE)从外显子层级定量信息和剪接读段中学习细胞嵌入表示。d, 在潜在空间中构建 K 近邻(KNN)图,以便根据多数投票原则从邻近细胞中优化并聚合剪接读段,从而提升下游剪接分析中的剪接覆盖度。e, 从聚合后的剪接读段中计算 percent-splice-in(PSI)值,使单细胞层面的可变剪接推断更加精确。f, DOLPHIN 生成的高分辨率细胞嵌入表示,能够比传统的基因表达量方法更好地刻画细胞异质性。g, 识别外显子特异性标志物。h, DOLPHIN 支持在多种细胞群体中开展广泛的可变剪接分析。
【结果
DOLPHIN 在多个真实单细胞数据集中展现出稳定且显著的性能提升,尤其体现在三个关键方面。首先,DOLPHIN 基于外显子结构建模的细胞嵌入表示比传统基因表达方法更加精细,能够更准确地区分细胞类型和亚型,提升了细胞聚类与可视化的分辨率。其次,得益于对外显子层级表达的建模,DOLPHIN 能够识别出传统方法难以检测的差异外显子标志物,揭示了在基因层面被掩盖的调控机制。最后,DOLPHIN 在单细胞层面支持精确的可变剪接(alternative splicing)分析,不仅显著提升了剪接事件的检测灵敏度,还揭示了多个具有功能意义的剪接模式,在疾病研究中展现出重要应用潜力。
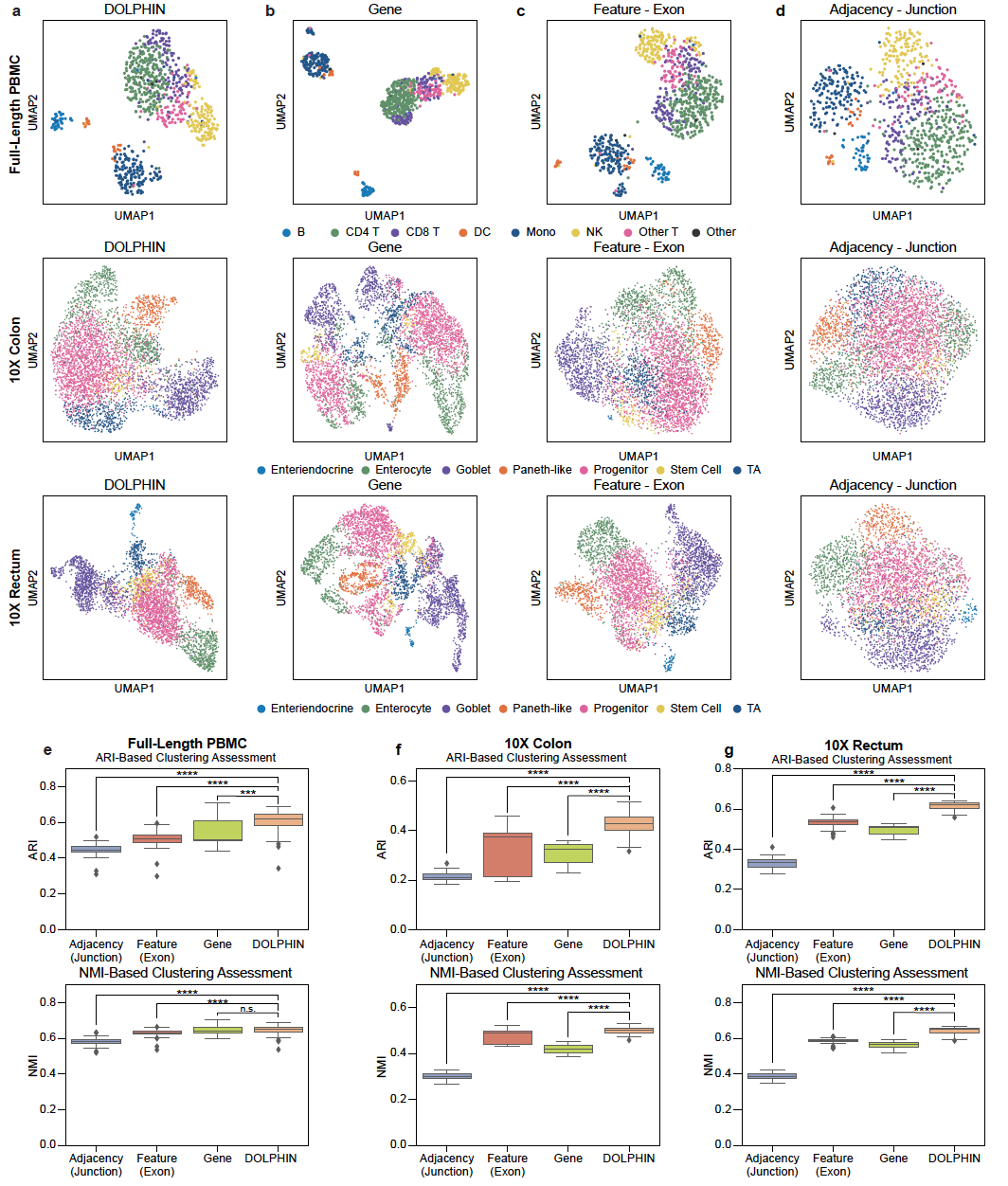
图2 DOLPHIN 通过整合外显子与剪接读段提升细胞嵌入质量。a–d, 不同单细胞 RNA 测序数据集中,使用 DOLPHIN 所生成的细胞嵌入(整合了外显子与剪接读段)与传统基因表达方法进行 UMAP 可视化对比。更清晰的细胞群体聚类和分离代表更高质量的嵌入。在每个数据集中,对以下输入进行比较:a, DOLPHIN 同时整合外显子与剪接读段,生成最具整合性和生物信息含量的细胞嵌入表示。b, 使用基因表达矩阵的 DOLPHIN 框架,代表传统基因层级分析。c, DOLPHIN 仅使用外显子读段(特征矩阵)。d, DOLPHIN 仅使用剪接读段(邻接矩阵)。e–g, 使用调整兰德指数(ARI)和归一化互信息(NMI)评分,对三种数据集中不同嵌入结果的聚类质量进行量化对比。
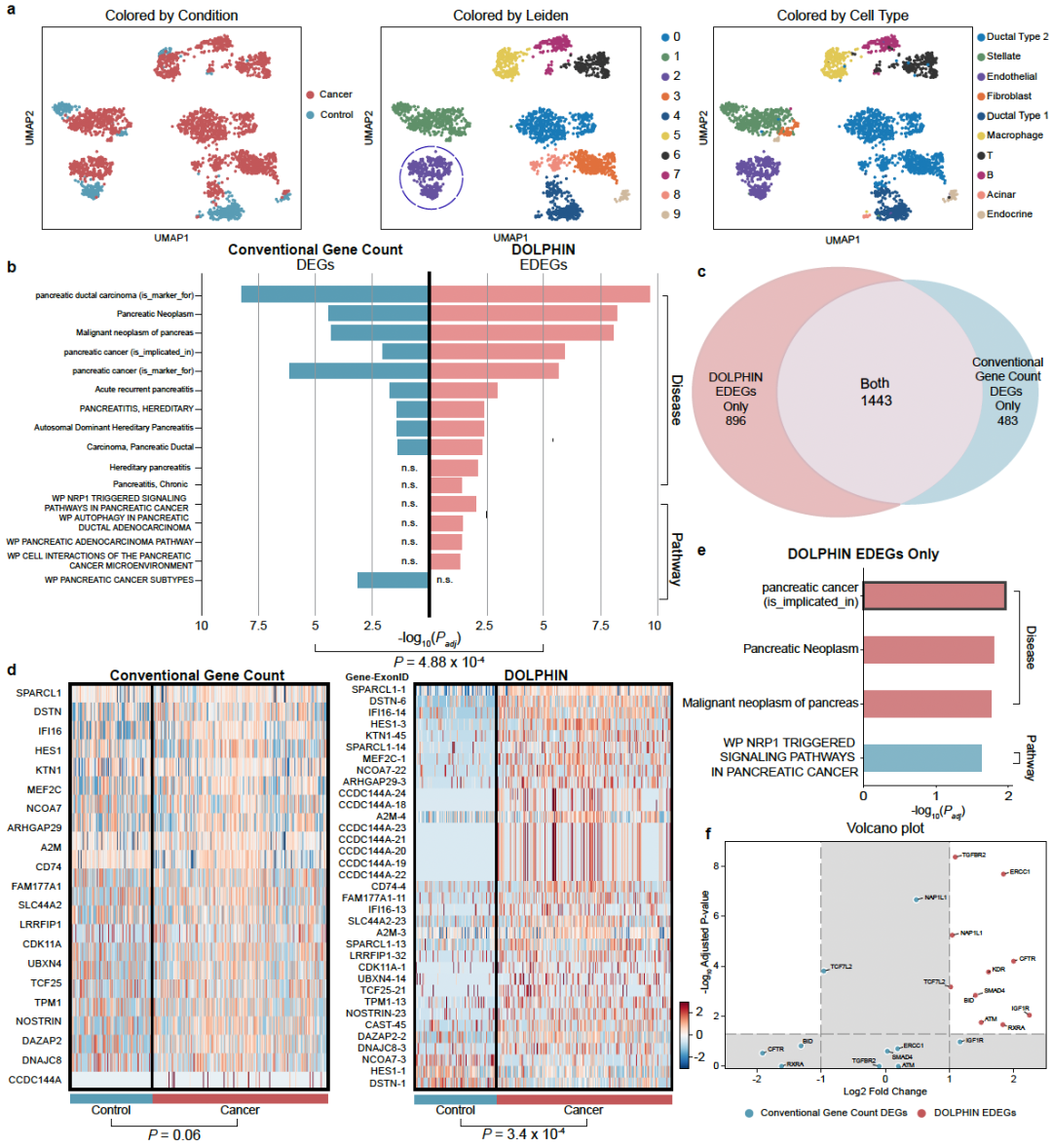
图3 DOLPHIN 能识别传统基因层级方法难以检测的外显子层级差异基因。a, 使用 DOLPHIN 对胰腺导管腺癌(PDAC)数据集进行聚类,被高亮的 Leiden cluster 2 被用于后续癌症与对照组的对比分析。b, 富集分析显示,DOLPHIN 识别出的外显子层级差异表达基因(EDEGs)在胰腺癌相关通路中具有更显著的富集程度,相比之下,传统基因层级差异表达基因(DEGs)的富集程度更弱,说明 DOLPHIN 能提供更深入的生物学洞察。c, Venn 图显示 DOLPHIN 识别出的 EDEGs 数量远多于传统方法检测出的 DEGs,突出其在识别生物学相关变异方面的敏感性。d, 热图展示了仅被 DOLPHIN 检测到的差异外显子在癌症与对照样本中的表达模式,同时显示对应基因层级表达。这说明 DOLPHIN 能捕捉基因层级无法识别的微妙转录变化。e, 对 896 个仅由 DOLPHIN 检测出的 EDEGs 进行富集分析,发现其显著关联于胰腺癌通路。而仅通过传统方法检测出的 483 个 DEGs 在这些通路中无显著富集。f, 火山图突出显示 DOLPHIN 从 e 部分提到的胰腺癌相关通路中识别出的 EDEGs。这些基因未被传统方法检测为 DEGs,显示 DOLPHIN 可挖掘出在基因层面被遗漏的关键外显子差异表达信号。灰色点表示差异不显著。
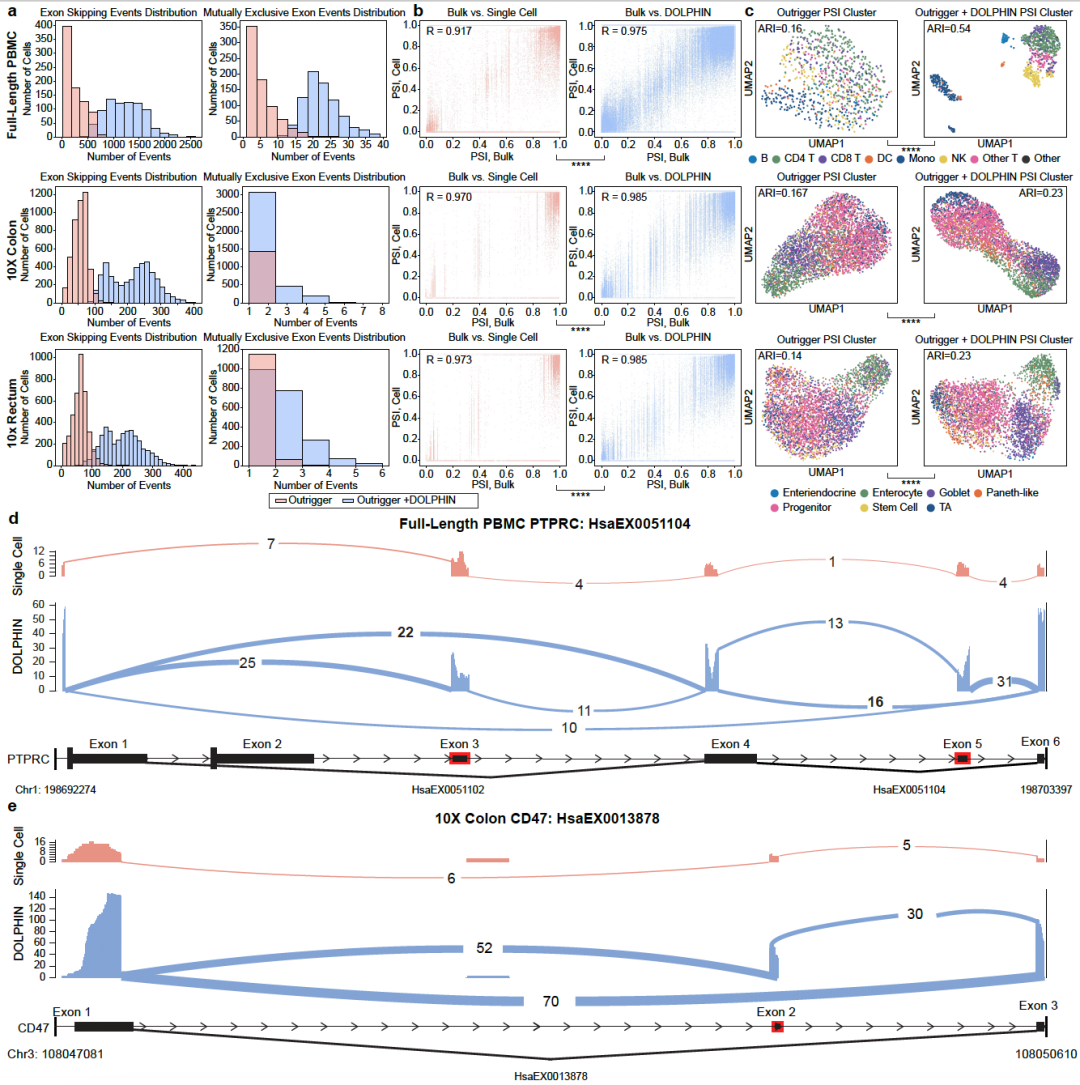
图4 DOLPHIN 增强了可变剪接事件的检测与分析能力。a–c, 在三个数据集中检测可变剪接事件(AS)。a, 与基线工具 Outrigger 相比,DOLPHIN 检测到更多的剪接事件。b, PSI 值的散点图显示,DOLPHIN 的剪接量化结果与伪 bulk 数据(作为参考标准)的相关性更高,说明其量化结果更准确。c, 基于 PSI 值的 UMAP 图揭示了 DOLPHIN 能捕捉到更加清晰、具有生物学意义的细胞类型特异剪接模式,提升了传统方法难以识别的剪接分辨率。d, 在全长 PBMC 数据集中,针对剪接事件 HsaEX0051104 的 Sashimi 图显示,DOLPHIN 聚合后产生更强的剪接读段信号,使原先被忽略的事件得以被检测到。e, 类似地,在 10X 结肠数据集中,DOLPHIN 对剪接事件 HsaEX0013878 的增强也揭示了传统方法漏检的 AS 事件。
总结与展望
DOLPHIN 在单细胞转录组学分析中引入了前所未有的外显子与剪接层级建模框架,为理解细胞间的剪接异质性和精细表达调控打开了新视野。通过整合外显子测序片段与剪接测序片段,DOLPHIN 能够在不增加实验成本的前提下,显著提升细胞表征质量、差异标志物检测能力和剪接事件的解析深度。该平台为肿瘤等复杂疾病研究提供了更加敏锐的工具,特别适用于探索亚型特异性剪接信号和外显子级生物标志物。这种高分辨率的细胞表征不仅揭示了传统方法无法捕捉的关键信息,也为构建更接近真实分子状态的虚拟细胞奠定了基础。借助高保真虚拟细胞,研究者可以在计算机中模拟疾病的发生与发展,识别关键分子事件,并探索个性化诊断和精准治疗的新路径。
参考资料
Song, K., Zheng, Y., Zhao, B. et al. DOLPHIN advances single-cell transcriptomics beyond gene level by leveraging exon and junction reads. Nat Commun 16, 6202 (2025).
https://doi.org/10.1038/s41467-025-61580-w
内容中包含的图片若涉及版权问题,请及时与我们联系删除

