——研究背景——
胆汁酸是肠道微生物调控宿主健康与代谢稳态的关键代谢物之一,是介导肠道微生物与宿主间信息交流的“密码”。虽然目前已经发现了近千种菌源胆汁酸类分子,但其合成途径大部分尚不清楚。胆汁酸代谢酶的发现与解析复杂耗时,且往往仅能从特定胆汁酸和产生菌出发识别相关代谢酶,缺乏高效的筛选策略。近年来,人工智能(AI)技术在酶的功能预测1和全新功能酶挖掘2方面取得了重要进展。如果能利用AI学习胆汁酸代谢酶所具备的特征,就有可能从大规模的蛋白序列数据中快速筛选出潜在的新型胆汁酸代谢酶,帮助实验高效解析胆汁酸的菌源合成途径。
2025年8月7日,北京大学化学与分子工程学院/前沿交叉学科研究院定量生物学中心/北大-清华生命科学联合中心来鲁华教授团队与医学部基础医学院姜长涛教授、药学院马明教授以及基础医学院汪锴研究员团队合作,在Cell杂志发表了题为Identification of gut microbial bile acid metabolic enzymes via an AI-assisted pipeline的研究论文,创造性地使用“底物口袋相似性”进行数据增强,开发了基于AI的胆酸代谢酶预测方法BEAUT及其交互式在线平台(https://beaut.bjmu.edu.cn/),在菌株和蛋白水平上验证了BEAUT发现胆汁酸合成酶的能力。基于BEAUT的预测结果,合作者通过实验发现了3-O-乙酰胆酸水解酶MABH、新骨架类型菌源胆汁酸3-乙酮脱氧胆酸(3-acetoDCA)及其代谢酶ADS,还发现3-乙酮脱氧胆酸在人群中广泛存在并具有重要的生理功能。
——研究内容——
开发预测菌源胆酸代谢酶的AI方法BEAUT
从大规模功能未知的蛋白序列数据中筛选具备特定功能的酶是一项具有广泛应用前景的任务。数据的匮乏是限制AI模型在该任务上表现的重要因素,因此设计合适的数据增强策略能显著提高模型在筛选特定功能酶时的泛化能力。此外,还需要注意到蛋白的序列数据远多于测定的结构数据,因此模型应注重利用序列信息;如果想筛选大规模序列数据库(百万至千万级别)则对模型的计算速度提出了要求。团队设计了一个基于全连接神经网络的二分类模型,基于蛋白序列输入预测其是否为胆汁酸代谢酶,并使用ESM-23蛋白质语言模型进行序列表示,使模型更容易学习序列中功能相关的信息。该模型的预测速度可满足至少百万级别的蛋白序列筛选需求。
在收集用于模型训练的正类样本时,团队注意到在UniProt中,能够代谢胆汁酸的酶在序列去冗余后仅有151个,难以训练常规的AI模型,需要数据增强。团队从PDB中收集了8个已知胆酸代谢酶与胆酸底物的复合物结构,使用本实验室开发的Cavity4工具提取底物活性口袋并用PocketMatch5工具分析相似性,发现不同的胆酸代谢酶尽管序列和结构有很大差异,但其底物活性口袋具有相似性。团队据此设计了基于底物口袋相似性的数据增强策略(图1)。首先,从151个已知胆酸代谢酶中通过Cavity提取得到85个有高质量结构的底物结合口袋;然后团队根据文献报道选择了7种胆酸代谢能力多样性强的肠道细菌基因组,使用ESMFold3预测了其中蛋白质的结构,再利用Cavity提取出用于比较的口袋。通过使用PocketMatch比较这些口袋与已知酶底物结合口袋的相似性,团队找到了2330个可能的胆酸代谢酶用于扩充模型训练所用的正样本。
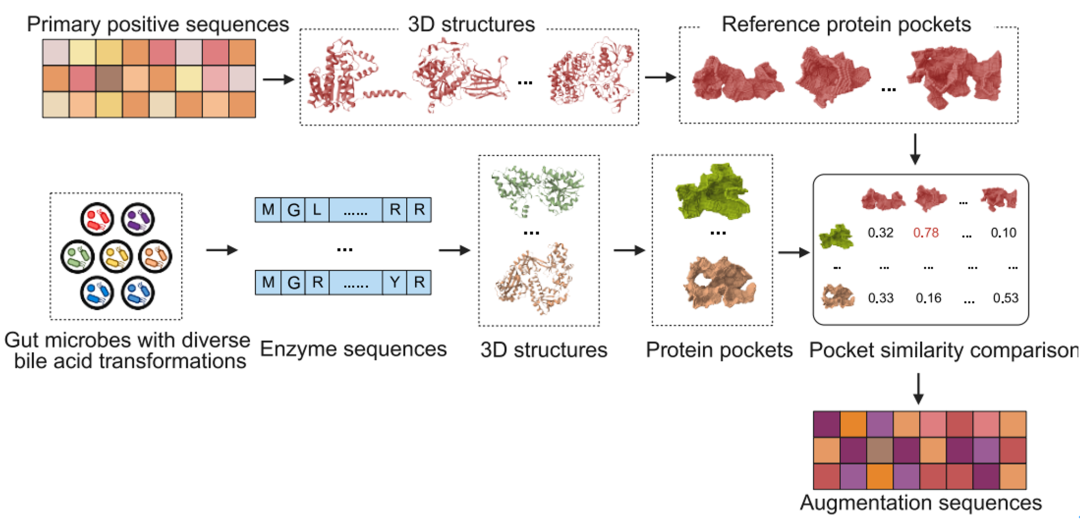
图1 BEAUT使用的数据增强策略
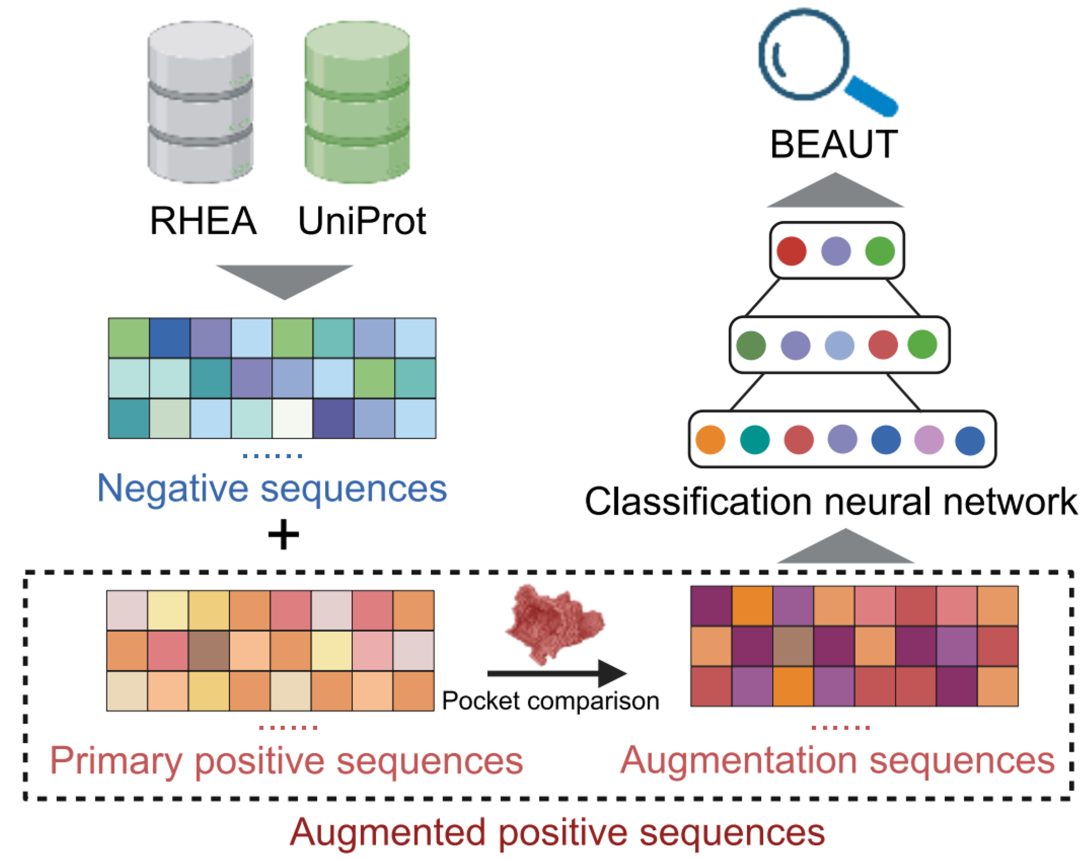
图2 BEAUT模型构建流程
使用扩充后的正样本结合适当的负样本数据,团队建立了基于序列的胆酸代谢酶预测模型BEAUT(图2)。为准确评估BEAUT的泛化能力,团队设计了与训练集序列同源性的独立测试集。在5折交叉验证中,BEAUT在该测试集上平均AUPRC达到0.80,平均F1-score为0.72,平均召回率为0.75. 这表明BEAUT能有效预测低同源性的潜在胆酸代谢酶。
BEAUT的实验验证
团队使用BEAUT对约230万条人体微生物基因组中包含的蛋白序列进行筛选,预测了约60万条潜在的胆汁酸代谢酶,团队将其整理在人体微生物胆汁酸代谢酶(HGBME)数据库中,数据库可在线访问(https://beaut.bjmu.edu.cn/database)。团队进一步评估了108株肠道细菌对胆汁酸的代谢能力,发现代谢能力与BEAUT预测的潜在代谢酶数量正相关。合作团队在预测结果中选择了102种酶进行实验验证,评估对于5种胆汁酸的代谢能力,发现其中有47种酶能够代谢至少一种胆汁酸(图3)。这表明BEAUT可以用于预测完全未知的胆汁酸代谢酶,可以大大提高对实验发现胆汁酸代谢酶的效率。
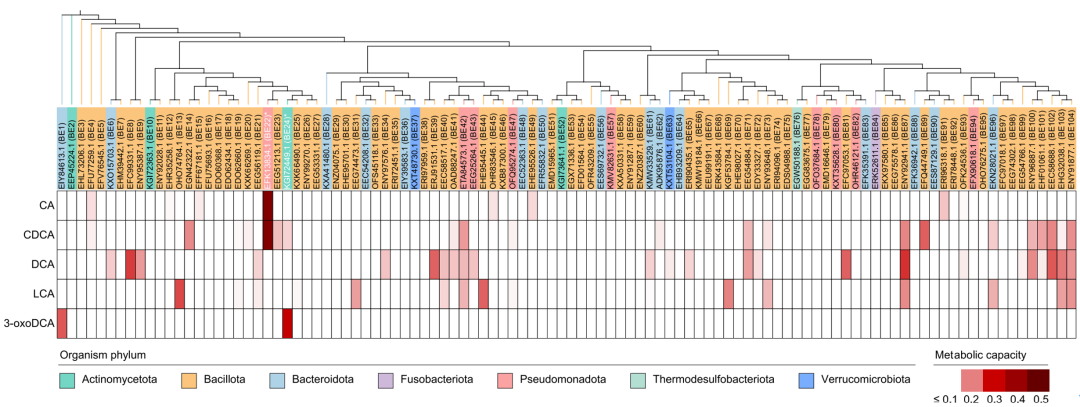
图3 BEAUT预测的102条潜在菌源胆汁酸代谢酶的实验验证结果
实验验证案例1:3-酰基胆酸水解酶MABH的发现
3-酰基胆酸是肠道FXR的强抑制剂,能调控宿主的代谢稳态,是潜在的抗代谢疾病分子,但其在人体内的代谢途径尚不清楚。团队使用BEAUT分析H. hathewayi基因组中的蛋白序列,首次发现了能水解3-乙酰胆酸的酶MABH(图4左)。MABH可能成为治疗代谢疾病的全新靶标。由于该酶在UniProt中被自动标注为Sialate O-acetylesterase家族成员,若使用常规方法则难以发现该酶代谢胆酸的功能。
实验验证案例2:3-乙酰脱氧胆酸合成酶ADS与3-乙酰脱氧胆酸的发现
合作团队使用BEAUT发现了有一类仅代谢3-oxoDCA的酶,且其代谢产物不符合经典的胆汁酸结构特征。经过鉴定,该酶催化丙酮酸与3-oxoDCA的缩合反应,生成3-乙酰脱氧胆酸(3-acetoDCA),这是胆汁酸被发现以来首次报道的全新骨架类型胆汁酸(图4右)。该胆汁酸的合成酶ADS也是一种全新功能的胆汁酸代谢酶,与已知的胆汁酸代谢酶序列和结构完全不同。3-acetoDCA不与经典的胆汁酸受体如FXR等等结合,但却能调控肠道共生菌Lactobacillus gasseri等乳杆菌的生长,并促进小鼠体内吲哚-3-乳酸的产生,介导了一类肠道菌群互作的新模式。该案例表明BEAUT可以突破已知胆汁酸代谢酶功能的限制,预测具有全新功能的胆汁酸代谢酶。
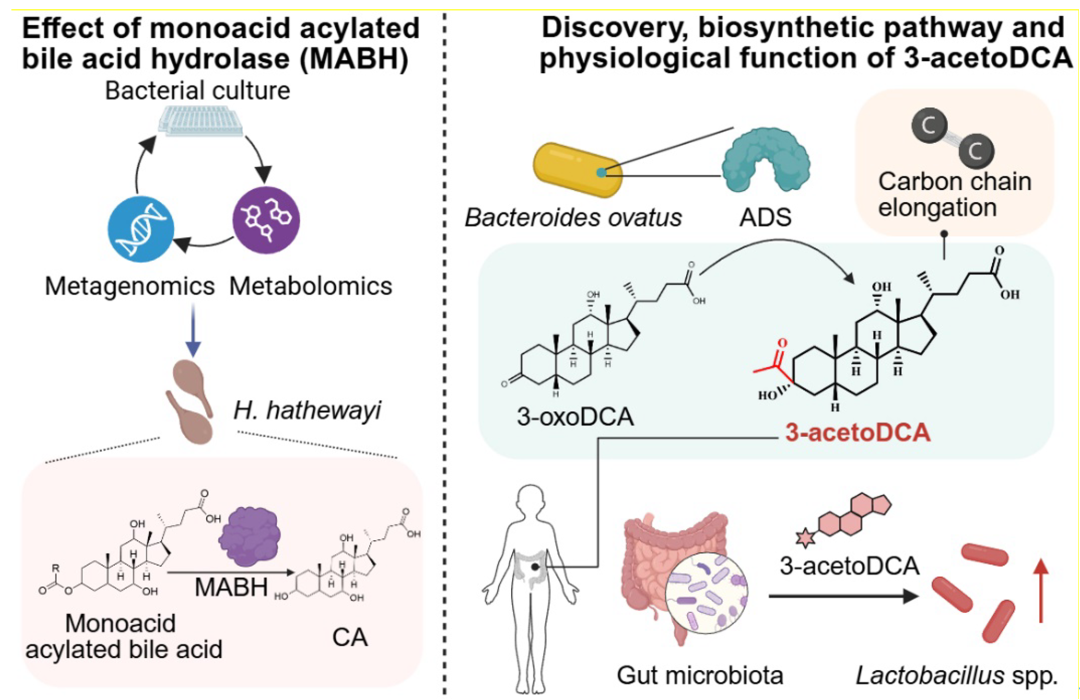
图4 BEAUT成功发现了菌源胆汁酸代谢酶MABH和ADS,和一种全新碳骨架胆汁酸3-acetoDCA
——小结——
胆汁酸的发现距今已过去100多年,但人们仍未能完全发现全部的胆汁酸类型及其代谢途径,胆汁酸的丰富生理功能还有待进一步解析。本工作创造性的引入“口袋相似性分析”解决了AI在小样本情景下的应用难题,成功构建了人体微生物菌源胆汁酸预测模型BEAUT,并预测了60余万条潜在的菌源胆酸代谢酶。BEAUT可通过交互式在线平台(https://beaut.bjmu.edu.cn/)供研究者使用。BEAUT的实验验证表明其可以有效预测具有全新功能的菌源胆汁酸代谢酶,并发现了3-酰基胆酸水解酶MABH和3-乙酮脱氧胆酸的合成酶ADS。BEAUT是人工智能与化学和生命科学研究结合的成功范例,将进一步推动人工智能在相关研究领域中的应用。
姜长涛教授、来鲁华教授、马明教授与汪锴研究员为本文的共同通讯作者。北京大学医学部基础医学院副研究员丁勇、博士后罗茜,化学与分子工程学院博士研究生郭家盛、药学院博士研究生邢佰颖以及前沿交叉学科研究院博士后林豪禹为本文的共同第一作者。来鲁华教授团队博士研究生郭家盛开发了BEAUT方法并整理了HGBME数据集,博士后林豪禹开发了BEAUT在线平台。本研究得到国家自然科学基金专项项目、重大研究计划集成项目、重点项目,以及国家重点研发计划项目等经费支持。
参考文献:
1.Yu, Tianhao, et al. "Enzyme Function Prediction Using Contrastive Learning." Science 379.6639 (2023): 1358-63. DOI: 10.1126/science.adf2465.
2.Huang, Jiaying, et al. "Discovery of Deaminase Functions by Structure-Based Protein Clustering." Cell 186.15 (2023): 3182-95. DOI: 10.1016/j.cell.2023.05.041.
3.Lin, Zeming, et al. "Evolutionary-Scale Prediction of Atomic-Level Protein Structure with a Language Model." Science 379.6637 (2023): 1123-30. DOI: 10.1126/science.ade2574.
4. Wang, Shiwei, et al. "Cavityplus 2022 Update: An Integrated Platform for Comprehensive Protein Cavity Detection and Property Analyses with User-Friendly Tools and Cavity Databases." Journal of Molecular Biology435.14 (2023): 168141. DOI: 10.1016/j.jmb.2023.168141.
5. Yeturu, Kalidas, and Nagasuma Chandra. "Pocketmatch: A New Algorithm to Compare Binding Sites in Protein Structures." BMC Bioinformatics 9.1 (2008): 543. DOI: 10.1186/1471-2105-9-543.

内容中包含的图片若涉及版权问题,请及时与我们联系删除

