结构域插入工程是一种将原本独立的生物功能进行空间连接的强大方法,可产生具有自然界中不存在活性的蛋白质。其中的一个突出例子是通过将受体结构域插入效应蛋白而构建的可切换蛋白变体。然而,找到适合的变构插入位点通常需要大量筛选和优化。研究人员提出了 ProDomino,一个基于机器学习的结构域重组设计流程,训练数据来自自然界中发生的结构域内插入事件并生成的半合成蛋白序列数据集。ProDomino 能稳健识别具有生物技术相关性的蛋白质中的插入位点,并在大肠杆菌和人类细胞中得到了实验验证。最后,研究人员使用光和化学可调控的受体结构域作为插入元件,快速、模型指导地构建了多种高效的单组分光遗传和化学遗传蛋白开关,包括可在人类细胞中进行诱导性基因组编辑的新型 CRISPR–Cas9 和 Cas12a 变体。本研究实现了一次性结构域插入工程,并显著加速了定制化变构蛋白的设计。

蛋白质的结构域是其结构和功能的基本单元。自然进化中,不同结构域组合的重新排列是新功能产生的重要来源。人工结构域重组已被证明是创造自然界中不存在的新功能蛋白的有力手段。为了不仅实现物理连接,还能有效耦合两个原本分离的蛋白功能,一个有效策略是将某一结构域插入到另一结构域的特定位点中,如果成功,就会产生结构和功能紧密依赖的融合蛋白。
这种策略在构建高灵敏生物传感器和新型分子机器方面具有巨大潜力,特别是在工程化变构蛋白开关方面。通过将光受体或配体结合结构域插入目标蛋白,可以获得其活性受光或小分子调控的可切换蛋白。然而,这一领域的进展受制于寻找合适插入位点的困难,因为随机的结构域融合往往破坏一个或两个结构域的结构与功能完整性。尽管表面暴露的柔性环区常被视为有前景的插入位点,但实验表明仅有少数此类环区能真正耐受插入。缺乏系统、可泛化的预测工具进一步限制了设计效率。
蛋白质语言模型的出现,为结构预测和功能预测带来了突破。然而,这些方法过去无法应用于结构域插入设计,原因在于缺乏适合的训练数据。研究人员的思路是,利用自然界中少见但存在的“结构域内插入”事件生成一个多样化的半合成数据集,以此训练能泛化到无关蛋白家族的预测模型。
结果
构建人工结构域插入数据集
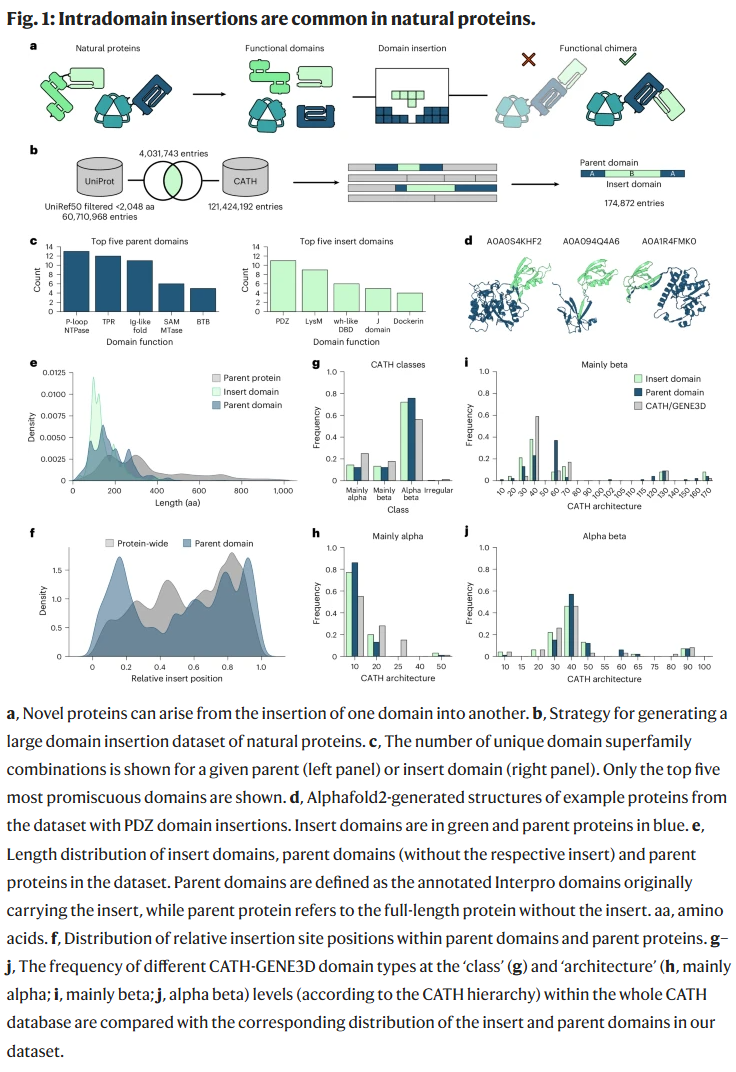
研究人员假设,自然界中发生的结构域内插入事件(一个结构域被另一个结构域“打断”并形成新结构域架构)能为结构域重组工程提供关键信息。相比常规的头尾拼接,这些插入更具结构限制性,也更可能携带功能耦合的信息。
为此,研究人员基于 CATH-Gene3D 结构域注释数据,筛选出一个结构域被另一结构域插入的蛋白,并通过 UniRef50 过滤同源性,最终获得 174,872 条来自不同生物的蛋白序列。这些数据涵盖了 202 种插入结构域超家族与 168 种母结构域类型,大多数结构域只作为插入或母体存在。插入位点在母结构域中呈双峰分布,中间插入的破坏性更大;在全长蛋白中则表现为 C 端偏向。分析显示,这种插入现象
在蛋白质世界中普遍存在,并不偏向特定结构域类别。
ProDomino 的训练与预测性能
基于该数据集,研究人员训练了多种模型,比较了不同编码方式(如 One-hot 与 ESM-2 表征)、不同数据划分方式(随机、基于结构域类别、严格单代表)及掩码策略的效果。结果表明,使用 ESM-2 表征、严格单代表划分及位置掩码能获得最佳泛化性能,模型被命名为 ProDomino。
在已知结构信息的蛋白中,ProDomino 没有表现出对特定二级结构或序列保守性的强偏好,但能正确识别实验验证的插入位点。例如,在 AraC 转录因子中,ProDomino 成功预测了两个已知光依赖调控的变构位点(I113、S170),并且性能优于先前基于梯度提升的模型。

在酶中识别变构插入位点
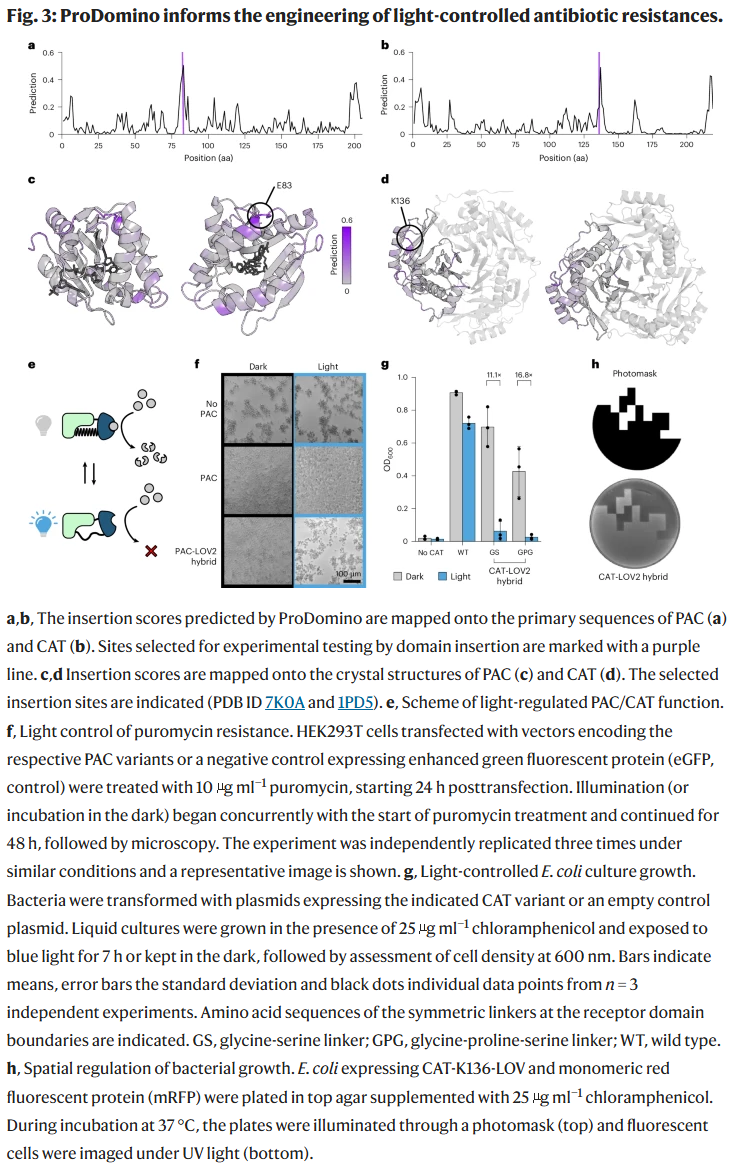
研究人员将 ProDomino 应用于常用的抗生素抗性酶——嘌呤霉素乙酰转移酶(PAC)和氯霉素乙酰转移酶(CAT)。模型预测的高分位点(PAC 的 E83 和 CAT 的 K136)位于表面环区。将 AsLOV2 光受体结构域插入这些位点后,得到的融合蛋白在黑暗中保留活性,光照下活性显著下降,实现了光控抗性功能。进一步验证显示,模型在 78% 的测试位点预测正确,尤其在高分位点的成功率更高。
构建光遗传与化学遗传的 CRISPR 编辑器
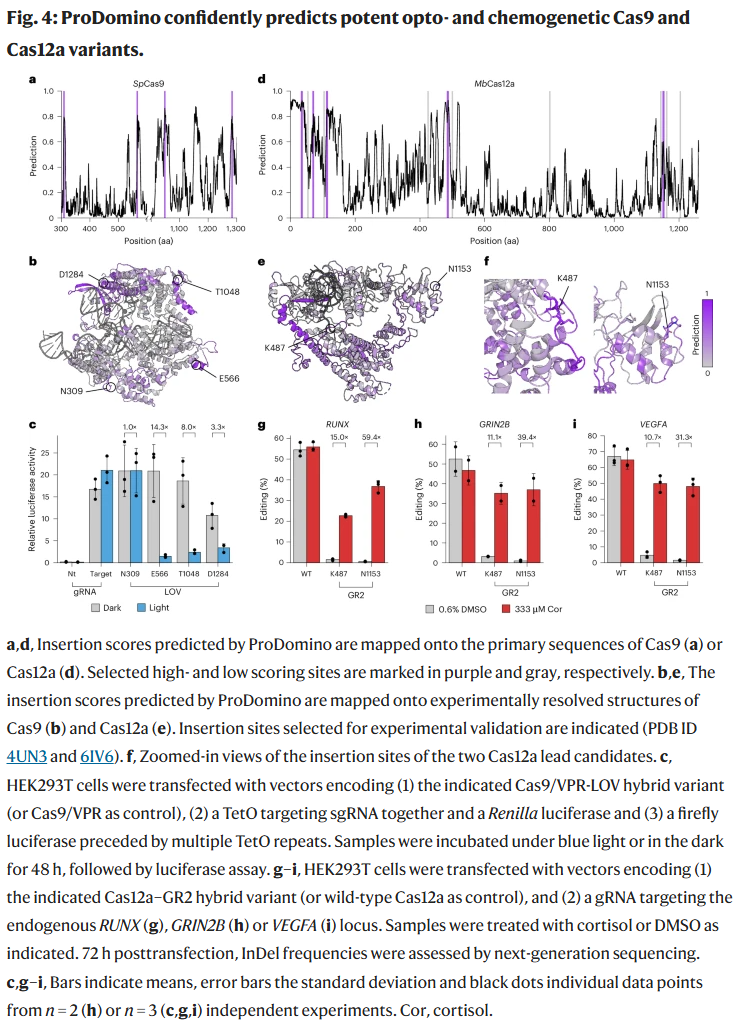
研究人员将 ProDomino 应用于复杂的多结构域蛋白 CRISPR–Cas9 和 Cas12a。对于 Cas9,模型与已报道的转座子插入数据有较高一致性,并识别出部分新位点,实验验证表明这些位点的 LOV2 插入可实现光依赖的转录激活调控。
对于 Moraxella bovoculi 来源的 Cas12a,模型预测其插入耐受性较广。研究人员在高分位点(K487、N1153)插入 LOV2 或化学受体 GR2,分别实现了光控和皮质醇依赖的基因组编辑。特别是 N1153-GR2 插入变体在有配体时编辑效率接近野生型,而无配体时几乎无活性,实现了高动态范围的化学调控。
讨论
研究人员提出的 ProDomino 模型依赖于一个全新的半合成结构域插入数据集,使其具备跨结构和进化关系泛化预测插入位点的能力。实验验证涵盖了来自不同结构与功能家族的多种蛋白,结果显示其预测准确性和在变构蛋白设计中的实用性。
在工程化可切换效应蛋白时,插入受体的选择、末端位置以及连接肽设计同样关键。研究人员在本研究中使用了小型对称连接肽(如 Gly-Ser、Gly-Pro-Ser),在插入耐受性与变构耦合之间取得平衡。未来可以结合结构建模工具进一步优化插入位点预测,并探索将 ProDomino 与人工设计的可切换受体结构域结合,用于全新功能蛋白的开发。
值得注意的是,从模型训练完成到所有可切换蛋白的克隆与验证,研究团队仅用了约 6 个月时间,这比以往的蛋白开关工程周期大大缩短。研究人员相信,ProDomino 将显著提高未来变构蛋白工程的效率和可定制化能力。
整理 | DrugOne团队
参考资料
Wolf, B., Shehu, P., Brenker, L. et al. Rational engineering of allosteric protein switches by in silico prediction of domain insertion sites. Nat Methods 22, 1698–1706 (2025).
https://doi.org/10.1038/s41592-025-02741-z

内容转自公众号【DrugOne】
内容中包含的图片若涉及版权问题,请及时与我们联系删除

