DRUGAI
今天为大家介绍的是来自纽约的纪念斯隆凯特琳癌症中心Wesley Tansey团队的一篇论文。大规模药物联合筛选(Combination Drug Screens)由于可能的组合数量庞大,通常被认为难以开展。现有方法通常采用固定的实验设计方案,然后利用机器学习模型来预测未测试的组合效果。本研究提出了一种创新方法BATCHIE,它采用分批次进行动态实验的策略。BATCHIE通过信息论和概率模型分析已有实验结果,从而优化设计后续批次的实验方案,以获取最大化的信息价值。在对已有大规模筛选实验数据的分析中,BATCHIE能够快速发现高效且具有协同效应的药物组合。研究团队在儿科癌症细胞系上对206种药物库进行前瞻性筛选时,BATCHIE仅需测试全部140万种可能组合中的4%,就能准确预测未知组合的效果并发现药物间的协同作用。更重要的是,该模型为尤文肉瘤找到了一系列最佳治疗组合,经后续实验验证确实有效,其中最具应用前景的是PARP抑制剂与拓扑异构酶I抑制剂的组合。研究结果表明,通过动态优化的实验设计,作者可以用相对较少的实验次数完成系统性的大规模药物联合筛选。BATCHIE已开源,可从https://github.com/tansey-lab/batchie获取使用。

在癌症、病毒和细菌感染的治疗中,单一药物治疗往往会产生选择压力,导致病原体产生耐药性,影响治疗效果。而联合治疗(Combination Therapies)能够同时阻断多个潜在的耐药途径,这也是为什么联合治疗在 HIV 等快速进化病原体的治疗中发挥重要作用,并逐渐成为抗生素和抗癌治疗的未来趋势。
然而,药物联合筛选面临着规模化的巨大挑战。随着实验条件的增加,可能的组合数量呈指数级增长。举例来说,对100种药物和50种细胞系在5个剂量下进行单药筛选只需2.5万次实验,但如果是两种药物联用,实验次数就会暴增至620万次。即使是最高效的高通量筛选团队,也难以完成如此庞大的实验工作。
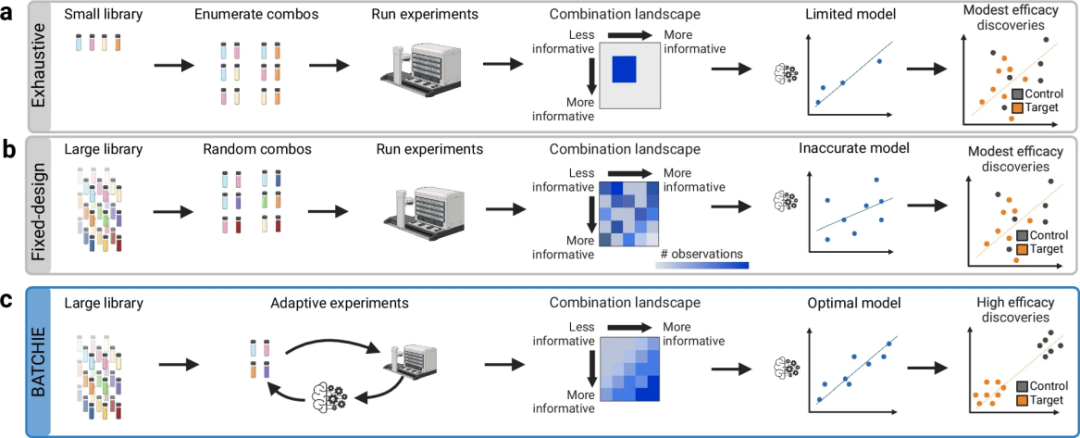
图 1
为解决这一难题,研究团队开发了BATCHIE系统。如图1所示,传统方法存在明显局限:
(1)使用规模较小的药物库可能完全找不到有效的组合;
(2)在大型药物库中采用随机或固定的筛选方案,往往会错过最有价值的组合。
BATCHIE采用创新的贝叶斯主动学习策略,通过动态优化实验设计来解决上述问题。该系统在尤文肉瘤研究中表现出色,成功发现了一系列具有高治疗效果比(治疗效果与副作用的比值)的药物组合。其中最具前景的组合是PARP 抑制剂与拓扑异构酶I抑制剂,该组合现已进入美国国家癌症研究所支持的II期临床试验。BATCHIE已开源发布,并提供了详细的使用教程。
联合药物筛选的适应性实验设计
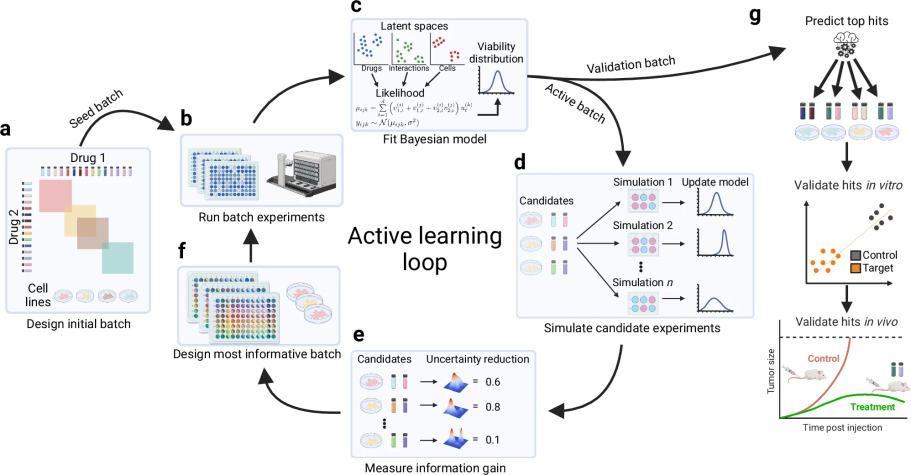
图 2
图2详细展示了BATCHIE系统如何通过智能化设计来最大限度地提高实验效率。整个流程可以分为以下步骤:
首先,系统会设计初始实验批次,确保对药物和细胞系进行全面且高效的覆盖(图2a)。完成这批实验后(图2b),系统利用实验数据训练一个智能预测模型,该模型能够预测不同细胞系对各种药物组合的反应(图2c)。
在后续批次中,BATCHIE会:
(1)利用已有数据预测新实验可能的结果(图2d);
(2)评估每个可能实验对提升预测准确度的贡献(图2e);
(3)设计最具价值的新实验批次(图2f)。
这个过程会不断循环,直到达到预设的实验次数或模型预测达到足够的准确度。最后,优化后的模型将用于预测最有希望的药物组合,并进行实验验证(图2g)。
BATCHIE采用了创新的概率直径主动学习(Probabilistic Diameter-based Active Learning, PDBAL)算法,这种算法特别适合处理药物筛选中的不确定性。它通过选择最有价值的实验来不断提高模型的预测准确度。在实际操作中,为了提高效率,实验采用特殊设计的微孔板进行。这种设计将药物分别放置在行板和列板中,通过组合可以测试所有可能的药物组合。这种创新的设计使得所需的实验板数量仅与药物数量呈线性增长,而不是与组合数量呈平方增长,大大提高了实验效率。
BATCHIE在现有组合数据集上的验证结果
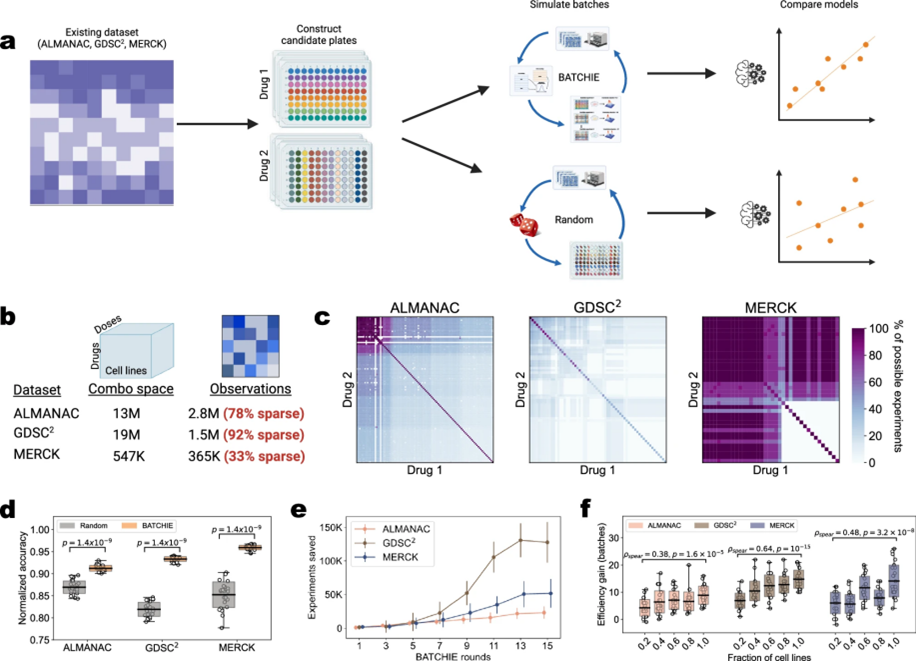
图 3
图3全面展示了BATCHIE系统的性能测试结果。研究团队首先设计了一个巧妙的验证方案(图3a):他们将现有的大型数据集重新组织成实验板的形式,这种设计与实际药物筛选实验中使用的方式完全一致。三个测试数据集(ALMANAC、GDSC2和MERCK)的数据分布各有特点(图3b-c),这种差异也为验证 BATCHIE 的通用性提供了良好的条件。测试结果令人印象深刻:
性能对比(图3d):BATCHIE 仅需15轮实验就达到了接近使用全部数据训练效果的表现,明显优于随机筛选策略;
效率提升(图3e):如果使用传统的随机策略要达到相同的效果,在不同数据集上需要额外进行数万次甚至十万次以上的实验;
扩展潜力(图3f):研究人员通过在不同规模的实验场景下进行测试,发现随着实验规模的扩大,BATCHIE 的优势会进一步放大。这一发现特别重要,因为它表明 BATCHIE 特别适合处理大规模的药物筛选任务。
这些结果不仅证明了BATCHIE的实用价值,更重要的是展示了它在提高大规模药物筛选效率方面的巨大潜力。通过智能化的实验设计,BATCHIE能够大幅减少所需的实验次数,同时保证发现有价值药物组合的能力。
儿科肉瘤探究与随机组合预测
研究团队选择儿科肉瘤作为BATCHIE系统的实际应用场景,这是因为许多儿科肉瘤,特别是转移性和复发性病例的治疗效果不尽如人意。图4全面呈现了BATCHIE系统在实际医学研究中的应用效果:
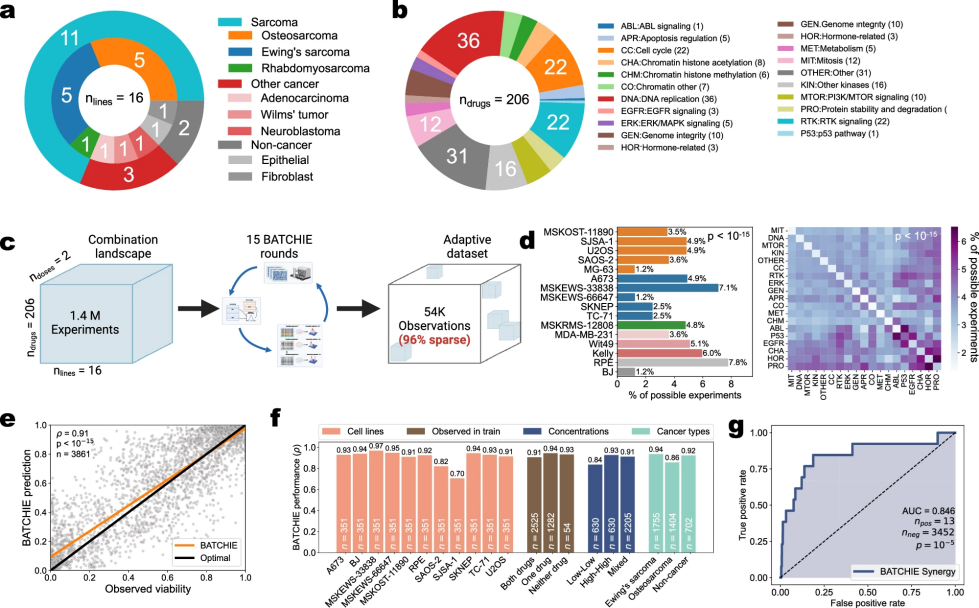
图 4
首先,研究团队精心设计了实验方案(图4a-b),选择了16种细胞系和206种药物进行测试,确保研究的全面性和可靠性。在实验过程中(图4c-d),BATCHIE 只需测试约4%的可能组合就获得了充分的信息。有趣的是,系统倾向于对某些细胞系和药物进行更频繁的测试,这反映了它能够识别出更有价值的实验组合。最令人印象深刻的是验证结果(图4e-g):模型在随机测试数据上展现出极高的准确性,且这种准确性在不同细胞系和药物剂量组合中都保持稳定。BATCHIE模型能够准确找出极其罕见的药物协同增效组合,这对开发新的联合治疗方案具有重要意义。
BATCHIE发现了治疗尤文肉瘤的合理药物组合
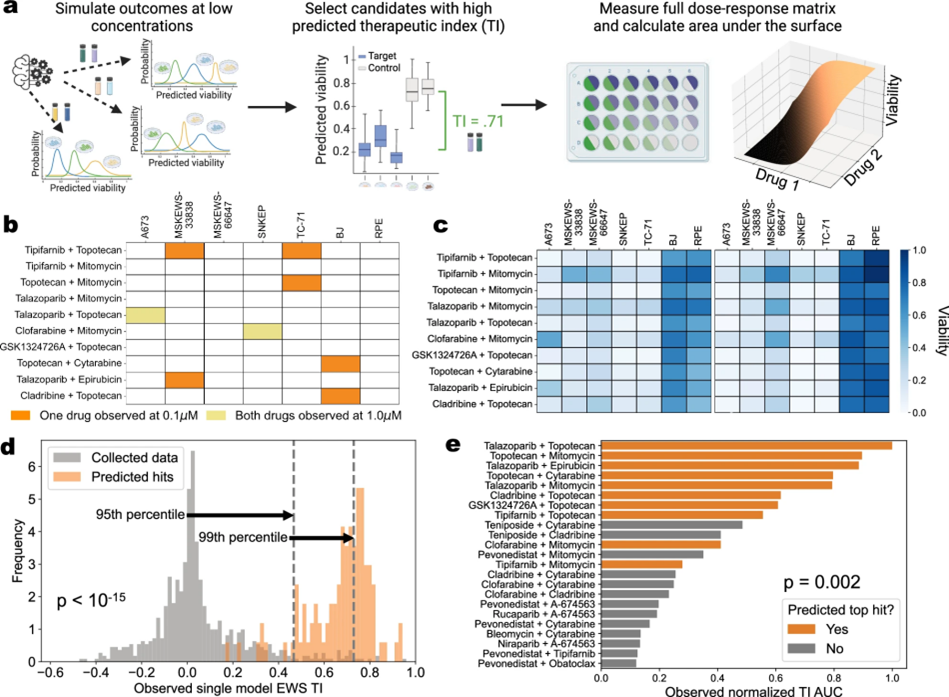
图 5
图5详细展示了BATCHIE系统如何帮助发现治疗尤文肉瘤的有效药物组合。研究团队首先利用系统预测筛选出最有潜力的药物组合(图5a)。为了确保结果可靠,他们不仅选择了10个最佳候选组合,还特意挑选了13个对照组合进行对比测试。验证结果令人振奋(图5b-e):
(1)BATCHIE对从未测试过的组合也能做出准确预测;
(2)发现的最佳组合效果远超常规组合,其治疗效果与副作用的平衡优于98%以上的已知组合;
(3)这些组合在不同剂量下都保持良好效果,显示出极强的稳定性。
特别值得一提的是,系统发现的Talazoparib和Topotecan组合已进入临床试验阶段。更重要的是,这些发现在患者样本中也得到验证,显示出从实验室到临床应用的巨大潜力。这些成果不仅证实了BATCHIE系统的实用价值,更为儿科肉瘤的治疗带来了新的希望。
BATCHIE发现了骨肉瘤与Aurora A 激酶抑制剂的相互作用
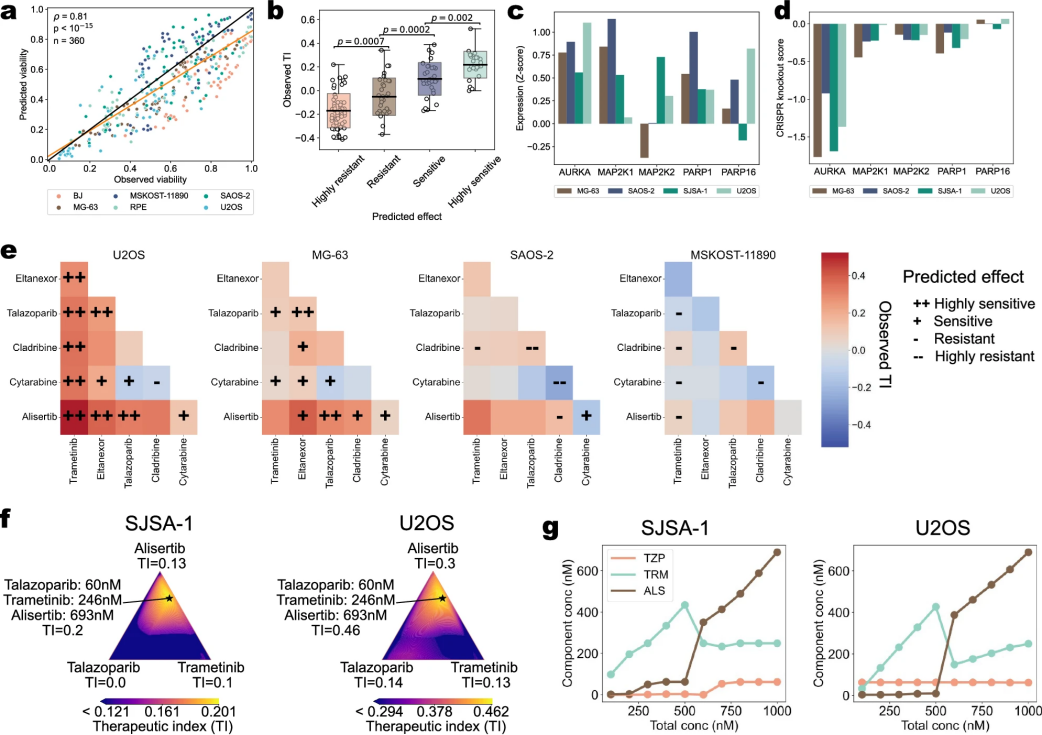
图 6
研究团队使用BATCHIE模型研究了骨肉瘤与药物组合的相互作用。与尤文氏肉瘤相比,骨肉瘤(OST)常发生染色体碎裂,导致每个肿瘤都具有独特的基因重排,因此具有更大的基因组多样性。图6展示了重要研究发现:
(1)模型预测的细胞存活率与实验观察结果高度一致(皮尔逊相关系数 = 0.81);
(2)Aurora A 激酶抑制剂alisertib、MEK抑制剂trametinib和PARP抑制剂talazoparib的组合在多个骨肉瘤细胞系(包括U2OS、MG-63和SAOS-2)中显示出高治疗指数(TI, 即药物的有效剂量与毒性剂量之比);
(3)通过DepMap数据分析发现,这些骨肉瘤细胞系都表现出AURKA(Aurora A 激酶)和MAP2K1/2(MEK激酶)的高表达,并对这些基因的敲除特别敏感;
(4)在三药联合实验中,以alisertib为主,配合低剂量trametinib和talazoparib的组合在部分骨肉瘤细胞中显示出最佳治疗效果。
由于alisertib在三期临床试验中失败而终止使用,研究团队还使用另一种在研的Aurora A激酶抑制剂LY329566进行验证,获得了相似的实验结果,为这一治疗策略提供了新的可能性。
结论
BATCHIE是一个基于主动学习的大规模药物组合筛选平台。研究表明,仅依靠生物学知识和专家经验难以设计最佳药物组合,因为疾病的异质性和药物的复杂作用机制。在GDSC2数据库中,药物与其作用机制类别外的相关性是类别内的两倍,且专家设计的组合方案在多数情况下效果欠佳。BATCHIE采用模块化设计,可整合各类贝叶斯模型,不仅适用于传统2D细胞系,还可扩展到3D模型和新兴技术领域,为开发新型组合疗法提供重要工具。
编译|于洲
审稿|王梓旭
参考资料
Tosh C, Tec M, White J B, et al. A Bayesian active learning platform for scalable combination drug screens[J]. Nature Communications, 2025, 16(1): 156.
内容中包含的图片若涉及版权问题,请及时与我们联系删除

